PCA3: Evidenz und klinische Bedeutung
Bussemakers et al. konnte 1999 das in Prostatatumoren deutlich überexprimierte DD3-Gen (später PCA3 genannt) identifizieren1. In der Folge bestätigte eine Vielzahl von Studien eine deutlich höhere PCA3-mRNA-Expression im malignen versus benignen Prostatagewebe2–7. Jedoch konnte der Großteil dieser Analysen keinen signifikanten Zusammenhang zwischen lokal fortgeschritten Stadien und Tumoraggressivität demonstrieren5–8.
In der Folge wurden drei verschiedene Test-Assays zur Messung von PCA3-mRNA aus dem Harn nach digital rektaler Untersuchung (DRU) entwickelt3, 9, 10. Aus den dabei in den Harn abgeschilferten Prostatazellen misst der Test PCA3-mRNA gemeinsam mit PSA-mRNA, welche bei neoplastischen Zellen im Gegensatz zur PCA3-mRNA nicht hochreguliert wird, um den Gehalt der prostataspezifischen mRNA im molekularen Testansatz zu normalisieren.
In diesem Review soll der aktuelle Stand der Evidenz im Zusammenhang mit der Rolle von PCA3 in Früherkennung, Active Surveillance (AS) und präoperativem Staging untersucht werden. Des Weiteren werden wichtige Aspekte wie klinische Anwendbarkeit von PCA3 und dessen Kombinierbarkeit mit anderen neuen Biomarkern diskutiert.
Entwicklung des PCA3-Tests
Das DD3PCA3– oder PCA3DD3-Gen, lokalisiert am Chromosom 9q21–22, wurde 1999 von Bussemakers et al. identifiziert und charakterisiert. Mittels „reverse transcriptase polymerase chain reaction“ (RT-PCR-Methode) demonstrierten die Autoren, dass PCA3 überexprimiert in Prostatakarzinomgewebe und niedrig exprimiert in nichtmalignen Prostatagewebe nachweisbar ist, aber nicht nachweisbar im gesunden Gewebe der Hoden, Niere, Samenblasen, Gehirn und Lunge1. De Kok et al. konnten nachweisen, dass die Expression von PCA3 mRNA 34-fach versus 6-fach höher bei malignen versus benignen Prostatagewebe war2. Basierend darauf wurde ein diagnostischer Test entwickelt. Hessels et al. berichteten als erste über die Messung von PCA3-mRNA in sedimentiertem Harn. Dabei wurde die erste Harnportion nach digital rektaler Untersuchung mit dreimal sanftem Abstreichen beider Prostataseitenlappen, von der Basis zum Apex, gewonnen und untersucht. Da die PSA-mRNA-Expression in normalen Prostatazellen konstant und in Karzinomzellen nur gering (1,5-fach) überreguliert ist, wurde der „PCA3 Score“ als Verhältnis zwischen PCA3-mRNA zu PSA-mRNA multipliziert mit 1.000 berechnet3. Unter Anwendung des PCA3-Tests (uPM3TM assay) der zweiten Generation konnten Tinzl et al.9 und Fradet et al.11 das gezeigte diagnostische Potenzial des Markers bestätigen. Im Jahre 2006 präsentierten Groskopf et al. einen völlig neuartigen PCA3-Assay, welcher sich durch gute Teststabilität und Anwendbarkeit im nichtsedimentierten Harn auszeichnet10. Diese Ergebnisse konnten in Folgestudien, welche die Rate an auswertbaren PCA3-Tests mit 94 bis 100 % belegten10, 12–20, bestätigt werden.
Zusammenfassend konnte mit PCA3 ein Test entwickelt werden, der im Jahr 2006 von der „Conformité européenne“ (CE) und im Jahr 2012 von der Food and Drug Administration (FDA) zertifiziert wurde und in der klinische Routine einfach anwendbar ist.
PCA3 in der Früherkennung
Aufgrund der bekannten Limitationen von PSA hinsichtlich seiner eingeschränkten Spezifität im Rahmen der Früherkennung zielte die Anwendung von PCA3 allen voran auf die Reduktion möglicherweise unnötiger Biopsien ab.
Als erste konnten Marks und Mitarbeiter in 226 konsekutiv rebiopsierten Männern die Überlegenheit von PCA3 gegenüber PSA (AUC = 0,68 vs. 0,52, p = 0,008) deutlich machen. Unter Anwendung eines Cut-off von 35 erzielte PCA3 eine Sensitivität, Spezifität und Odds Ratio von 58 %, 72 % und 3,614. In der Folge wurden prospektive, multizentrische Studien in den USA und Europa aufgelegt, in welchen der PCA3-Score eine Vorhersagegenauigkeit (AUC) eines Karzinoms in der Erst- oder Rebiopsie von bis zu 68 % erzielte21, 13. In einer Subgruppenanalyse (n = 301) der europäischen Multicenterstudie konnten Ploussard et al. die Überlegenheit von PCA3 gegenüber %fPSA als Prädiktor für eine positive Biopsie darstellen (AUC = 0,69 vs. 0,57)22. In einer weiteren Studie wurde auch die Überlegenheit von PCA3 gegenüber PSA-velocity demonstriert, wenngleich der neue Marker in der dritten Rebiopsie nur mehr geringe diagnostische Vorteile gegenüber den etablierten Risikofaktoren aufwies23.
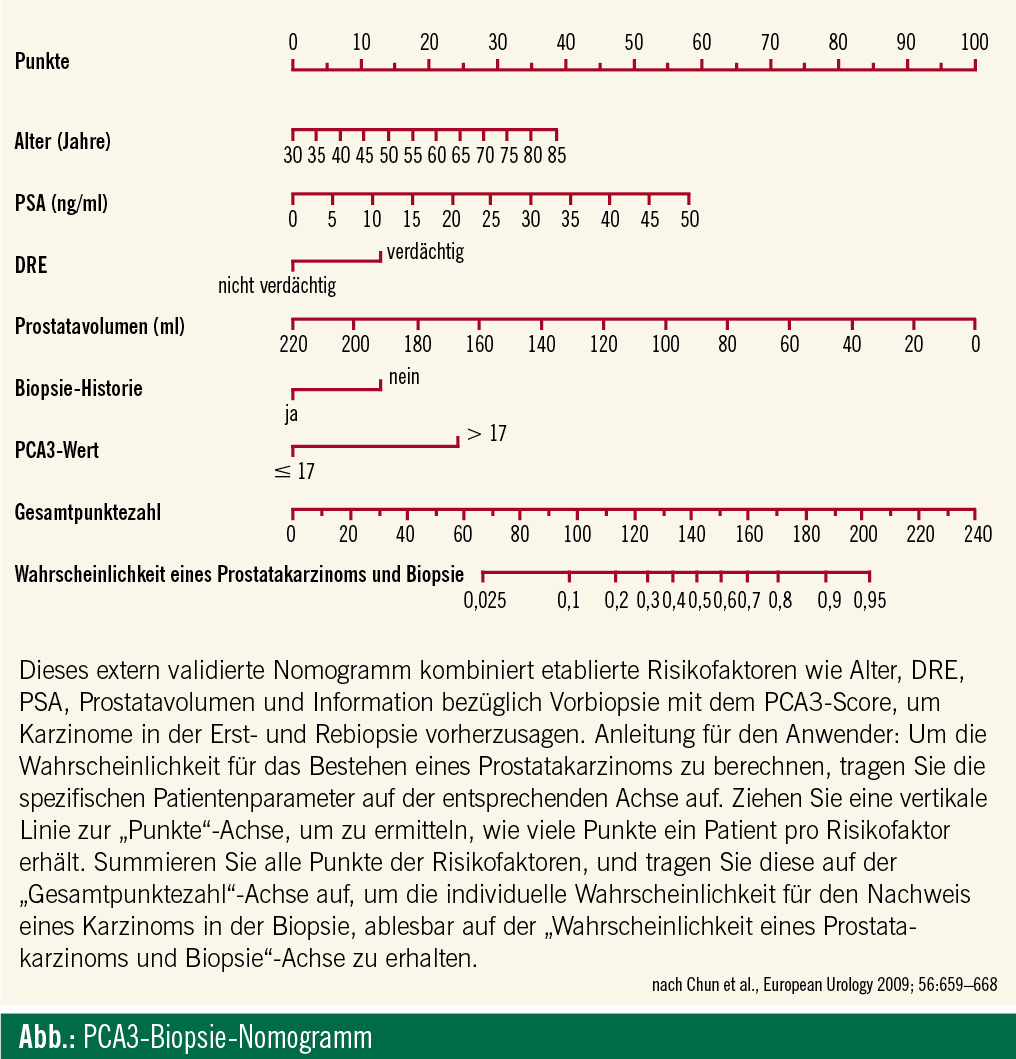
In der Folge konnten Chun et al. PCA3 als klinisch wertvollen, unabhängigen Prädiktor in einer großen US-europäischen Erst-und Rebiopsiekohorte bestätigen, welcher wie auch in der externen Validierung24, die Vorhersagegenauigkeit des Basismodells (Alter, PSA, DRE, Prostatavolumen und Biopsie-Historie; Abb.) um bis zu 5 %25 verbesserte.
Zusammenfassend konnte PCA3 als verlässlicher Prädiktor einer positiven Biopsie bestätigt werden. Die kombinierte Anwendung von PCA3 gemeinsam mit etablierten Risikofaktoren ermöglicht eine sichere und verbesserte Risikoeinschätzung, welche für den Kliniker in der Beratung von Patienten anwendbar wäre.
Anwendbarkeit von PCA3 bei AS und Staging
In einer einzigen Studie wurde PCA3 hinsichtlich seiner Anwendbarkeit bei klinisch signifikanten Tumoren im AS-Setting untersucht. Dabei wurde ein Trend zu höheren PCA3-Werten bei Patienten mit Gleason-Score-Upgrade in der Folgebiopsie nachgewiesen (72 vs. 50,8; p = 0,08)17. Da PCA3 im überwiegenden Anteil der Studien nicht als verlässlicher Marker der Tumoraggressivität bestätigt werden konnte2, 3, 14, 21, muss diese Rolle in größeren Serien analysiert werden.
Anschließend daran wurde der Zusammenhang des PCA3-Werts mit dem pathologischen Tumorvolumen, -stadium und der -aggressivität untersucht. Nakanishi und Mitarbeiter demonstrierten die Fähigkeit von PCA3, kleine Tumoren (Tumorvolumen < 0,5 ml) unabhängig vorherzusagen26. Whitman et al. zeigten als einzige auch die Vorhersage von extrakapsulärem Wachstum durch PCA327. Gils et al. und Hessels et al. konnten keinen dieser Zusammenhänge nachweisen28, 29. Die bisher größte publizierte Serie (n = 305) zeigte eine Verbesserung der multivariablen AUC für kleinvolumige Tumoren und pathologisch insignifikante Prostatakarzinommodelle, wenn PCA3 mit etablierten Risikofaktoren kombiniert wurde. Anderseits wurde keine Korrelation zu extrakapsulärem Wachstum und eine nur eingeschränkte Signifikanz zur Tumoraggressivität nachgewiesen30. Vergleichbare Ergebnisse zeigte eine Studie mit 106 Männern, welche sich bei klinisch geringer Risikokonstellation einer radikalen Prostatektomie unterzogen. Niedrige PCA3-Werte und günstige Biopsiekriterien (< 33 % oder 3 mm Tumor, < 3 positive Zylinder) sagten unabhängig kleine (Tumorvolumen < 0,5 ml) und insignifikante Prostatakarzinome vorher31.
Zusammenfassend kann PCA3 unter Vorbehalt der relativ kleinen Gesamtpatientenanzahl (n = 722) in Kombination mit etablierten Risikofaktoren zur Vorhersage kleiner und pathologisch insignifikanter Prostatakarzinome angewendet werden.
Kombination von PCA3 mit neuen Biomarkern
Nachdem PCA3 als hochspezifischer und klinisch praktikabler Prostatakarzinommarker vorgestellt wurde, könnten Kombinationen mit neuen Markern dieses Potenzial weiter verbessern. Dazu wurde im post DRE-Harn von 108 Biopsiepatienten die Fusion zwischen dem Transmembran-Serin-Protease-Gen (TMPRSS2) und dem „v-ets erythroblastosis virus E26 Oncogen“ (ERG) in Kombination mit PCA3 getestet. Interessanterweise wurde diese spezifische Fusion nur in 59 % des untersuchten Prostatagewebes gefunden. Die Kombination beider Marker erzielte eine Sensitivität und Spezifität von 73 % und 63 %32. In einer gemischten Biopsiekohorte (Initialbiopsie, AS-Folgebiopsie) mit 291 Patienten konnte Cornu et al. durch eine Kombination von TMPRSS2-ERG-Fusion, PCA3 und PSA-Density eine signifikante Verbesserung der Vorhersagegenauigkeit (AUC = 0,743) erzielen33.
Ähnliche Zugewinne an diagnostischer Genauigkeit konnte Laxman und Mitarbeiter durch Kombination von PCA3 mit GOLPH2, SPINK1 und TMPRSS2-ERG-Fusion34 und Ouyang et al. mit PCA3 und „prostate-specific G-protein coupled receptor“ (PSGR) erzielen35.
Gemeinsam mit PCA3 wurde eine neue PSA-Isoform, [−2]proPSA, in 160 Männern vor Erstbiopsie untersucht. Die Kombination der beiden neuen Marker verbesserte die multivariable Vorhersagegenauigkeit auf AUC = 0,77 und erzielt in der Decision-Curve-Analyse den höchsten Net-Benefit36.
Unter dem Vorbehalt einer Bestätigung durch weitere Studien (die Patientenzahl ist derzeit noch deutlich limitiert) könnten diese neuen Marker in naher Zukunft die Vorhersagefähigkeit etablierter Risikomodelle verbessern.
1 Bussemakers MJ et al., Cancer Res 1999; 59:5975–9.
2 de Kok JB et al., Cancer Res 2002; 62:2695–8.
3 Hessels D et al., Eur Urol 2003; 44:8–15; discussion 15–6.
4 Landers KA et al., Int J Cancer 2005; 114:950–6.
5 Schmidt U et al., Prostate 2006; 66:1521–34.
6 Bialkowska-Hobrzanska H et al., Can J Urol 2006; 13:2967–74.
7 Popa I et al., Mod Pathol 2007; 20:1121–7.
8 Klecka J et al., Anticancer Res 2010; 30:665–70.
9 Tinzl M et al., Eur Urol 2004; 46:182–6; discussion 87.
10 Groskopf J et al., Clin Chem 2006; 52:1089–95.
11 Fradet Y et al., Urology 2004; 64:311–5; discussion 15–6.
12 Schilling D et al., Urol Int 2010; 85:159–65.
13 Haese A et al., Eur Urol 2008; 54:1081–8.
14 Marks LS et al., Urology 2007; 69:532–5.
15 Sokoll LJ et al., Clin Chim Acta 2008; 389:1–6.
16 Shappell SB et al., Urology 2009; 73:363–8.
17 Tosoian JJ et al., J Urol 2010; 183:534–38.
18 Wang R et al., Cancer 2009; 115:3879–86.
19 Nyberg M et al., Scand J Urol Nephrol 2010; 44:378–83.
20 Aubin SM et al., J Urol 2010; 184:1947–52.
21 Deras IL et al., J Urol 2008; 179:1587–92.
22 Ploussard G et al., BJU Int 2010; 106:1143–47.
23 Auprich M et al., BJU Int 2011; 109:1627–35.
24 Auprich M et al., Eur Urol 2010; 58:727–32.
25 Chun FK et al., Eur Urol 2009; 56:659–68.
26 Nakanishi H et al., J Urol 2008; 179:1804–9; discussion 09–10.
27 Whitman EJ et al., J Urol 2008; 180:1975–8; discussion 78–9.
28 Hessels D et al., Prostate 2010; 70:10–6.
29 van Gils MP et al., Prostate 2008; 68:1215–22.
30 Auprich M et al., Eur Urol 2011; 59:96–105.
31 Ploussard G et al., Eur Urol 2011; 59:422–29.
32 Hessels D et al., Clin Cancer Res 2007; 13:5103–8.
33 Cornu JNet al., Prostate 2012; epub ahead of print
34 Laxman B et al., Cancer Res 2008; 68:645–9.
35 Rigau M et al., Prostate 2010; 70:1760–67.
36 Perdona S et al., Prostate 2012; epub ahead of print