Künstliche Intelligenz in der (Immun)Pathologie
Die Integration von künstlicher Intelligenz (KI) in die Pathologie stellt einen grundlegenden Wandel im diagnostischen Spektrum der Onkologie dar. Basierend auf der zunehmenden Digitalisierung histologischer Präparate mittels Whole Slide Imaging (WSI) eröffnen sich neue Möglichkeiten zur algorithmischen Analyse großer Bilddatensätze.
Diese Entwicklung ermöglicht nicht nur eine standardisierte und reproduzierbare Bewertung histologischer Strukturen, sondern schafft auch die Grundlage für die computergestützte Vorhersage molekularer Alterationen und die quantitative Analyse des Tumormikroenvironments. Insbesondere in der Immunonkologie, wo immunhistochemische Marker und zelluläre Kontextanalysen entscheidend für Therapieentscheidungen sind, bietet KI ein vielversprechendes Potenzial zur Erweiterung des diagnostischen Repertoires.
Durch die Etablierung KI-gestützter Workflows können relevante histopathologische, immunologische und molekulare Informationen zunehmend automatisiert erfasst, integriert und in klinische Entscheidungsprozesse eingebunden werden. Damit wird KI zu einem Schlüssel-instrument für eine präzisere und datenbasierte Onkologie.
Fundamentale Entwicklungen: Digitale Pathologie und KI
Basis: Die technologische Basis für KI-gestützte Anwendungen in der Pathologie ist die Digitalisierung histologischer Präparate, insbesondere durch sogenannte Whole Slide Imaging (WSI)-Systeme. Diese ermöglichen es, komplette Gewebeschnitte in hochauflösende digitale Bilder umzuwandeln, die anschließend archiviert, geteilt und automatisiert ausgewertet werden können. Die Einführung von WSI markiert damit einen entscheidenden Schritt in Richtung einer datengetriebenen, rechnergestützten Pathologie.
Konzept der digitalen Pathologie: In der Folge hat sich das Konzept der digitalen Pathologie etabliert – also der Übergang vom klassischen Lichtmikroskop zur Bildanalyse am Bildschirm, ergänzt durch Softwarelösungen für Annotation, Fallverwaltung und Befunderstellung. Diese Digitalisierung schafft die Voraussetzungen für den Einsatz künstlicher Intelligenz, insbesondere von Deep-Learning-Verfahren wie Convolutional Neural Networks (CNNs) (Abb.) oder Foundation-Modellen, die auf der automatisierten Erkennung visueller Muster in Bilddaten beruhen.1–3
CNNs sind in der Lage, komplexe histologische Merkmale zu identifizieren, zu klassifizieren und quantitativ auszuwerten – mit einem Maß an Präzision, das je nach Anwendungsbereich menschlicher Beurteilung ebenbürtig oder sogar überlegen sein kann. Durch Training auf großen, annotierten Bilddatensätzen lernen diese Modelle, relevante Strukturen zu unterscheiden – z.B. Tumor vs. Normalgewebe, Grad der Differenzierung, das Vorhandensein bestimmter Zelltypen oder pathologische Veränderungen im Stroma.4
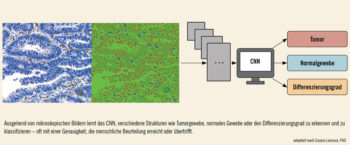
Abb.: Illustratives Beispiel, wie Convolutional Neural Networks (CNNs) genutzt werden, um histologische Gewebeproben zu analysieren.
Einsatz von KI-Tools: In der gegenwärtigen Praxis kommen KI-Tools v.a. als Assistenzsysteme zum Einsatz. Ihr Ziel ist es, Patholog:innen bei wiederkehrenden oder besonders komplexen Aufgaben zu unterstützen – etwa bei der standardisierten Quantifizierung von Proliferationsmarkern wie Ki-67 oder der Beurteilung immunhistochemischer Färbungen (z.B. PD-L1, HER2). Studien zeigen, dass KI in der Lage ist, die Inter-Observer-Variabilität bei solchen Beurteilungen deutlich zu reduzieren und so zu einer erhöhten diagnostischen Genauigkeit beizutragen.5–7
Darüber hinaus können KI-basierte Systeme Arbeitsabläufe beschleunigen, indem sie z.B. auffällige Bildbereiche automatisch markieren oder Fälle mit hoher Relevanz für die menschliche Nachkontrolle priorisieren. In der Breite steht die Anwendung von KI in der Pathologie jedoch noch am Anfang: technische Herausforderungen (z.B. Variabilität in der Gewebeaufbereitung, Scannerartefakte) sowie die Notwendigkeit regulatorischer Validierung und klinischer Integration begrenzen derzeit den Routineeinsatz.8
Immunpathologie – Neue Einblicke in das Tumormikroenvironment
Die Analyse des Tumormikroenvironments (TME) ist ein zentrales Element der modernen onkologischen Diagnostik, insbesondere im Hinblick auf das Ansprechen auf Immuntherapien. Die Immunpathologie befasst sich dabei mit der Charakterisierung immunologischer Zellpopulationen im Tumorgewebe, wie z.B. tumorinfiltrierender Lymphozyten (TILs), Makrophagen, dendritischer Zellen oder regulatorischer T-Zellen. Ihre Verteilung, Dichte und räumliche Organisation sind entscheidende Parameter für die Prognoseabschätzung und Therapieentscheidung, etwa bei Checkpoint-Inhibitor-basierten Therapien.
Künstliche Intelligenz – insbesondere auf Deep Learning basierende Bildanalyseverfahren – ermöglicht es, diese komplexen zellulären Strukturen automatisiert, reproduzierbar und quantitativ zu erfassen. Mittels segmentierender und klassifizierender Algorithmen können histologische Bilddaten analysiert und immunrelevante Zelltypen identifiziert und gezählt werden. Dabei gelingt es KI-Systemen zunehmend, zwischen unterschiedlichen Immunzellsubtypen zu differenzieren, auch wenn diese morphologisch nur schwer zu unterscheiden sind. Beispiele hierfür sind die Unterscheidung zwischen zytotoxischen und regulatorischen T-Zellen oder M1- vs. M2-Makrophagen anhand von Färbemustern und Zellmorphologie.9, 10
Räumliche Analyse des Tumor-Immunsystems: Ein besonders spannendes Feld ist die räumliche Analyse des Tumor-Immunsystems. KI kann nicht nur Zelltypen erkennen, sondern auch deren räumliche Verteilung innerhalb der Tumorarchitektur analysieren – etwa im Zentrum des Tumors vs. Invasionsrand oder in Relation zu tumorassoziierten Stromazellen. Solche Analysen erlauben Rückschlüsse auf Immunescape-Mechanismen, die für die Therapieantwort von zentraler Bedeutung sind. Neuere Studien zeigen, dass z.B. bei Darmkrebs und Melanom bestimmte räumliche Muster der Immunzellinfiltration – wie eine hohe Dichte an TILs im peripheren Invasionsbereich – mit einer besseren Prognose korrelieren.11, 12
Langfristiges Ziel: Entwicklung eines „digitalen Immunprofils“: Darüber hinaus eröffnet die Kombination von KI-gestützter Bildanalyse mit molekularbiologischen Methoden neue Perspektiven. Durch die Integration von spatial transcriptomics, single-cell RNA-Seq und proteo-mischen Daten lassen sich multimodale KI-Modelle trainieren, die zelluläre Interaktionen, Signalnetzwerke und funktionelle Immunantworten innerhalb des Tumorgewebes modellieren können.13 Ziel ist eine holistische Charakterisierung des Immunmikroenvironments, die sowohl strukturelle als auch funktionelle Parameter berücksichtigt.
In präklinischen Studien konnten solche multimodalen Ansätze bereits genutzt werden, um Immunphänotypen zu klassifizieren, Subgruppen mit therapeutisch relevantem Immunsignaturprofil zu identifizieren und potenzielle Biomarker-basierte Responder-Profile für Immuncheckpoint-Inhibitoren zu definieren.14, 15
Langfristig könnte KI auf dieser Basis zur Entwicklung eines „digitalen Immunprofils“ beitragen, das sowohl für die Primärdiagnostik als auch für die Therapieplanung und Verlaufsbeurteilung genutzt werden kann – insbesondere bei immunmodulatorischen Therapien im onkologischen Kontext.
KI-gestützte Prädiktion molekularer Marker
Ein zunehmend dynamisches Forschungsfeld innerhalb der digitalen Pathologie ist die Prädiktion molekularer Tumorcharakteristiken direkt aus morphologischen Bilddaten ohne zusätzliche molekularbiologische Tests. Solche Modelle können subtile histologische Muster erkennen, die mit genetischen, epigenetischen oder transkriptionellen Veränderungen korrelieren.
Zahlreiche Studien konnten zeigen, dass CNNs in der Lage sind, klinisch relevante molekulare Alterationen aus digitalen H&E-Bildern mit hoher Genauigkeit vorherzusagen.
Dazu zählen beispielsweise:
- Mikrosatelliteninstabilität (MSI) bei kolorektalem und Endometriumkarzinom16
- Tumormutationslast (TMB) als Marker für Immuntherapieansprechen17, 18
- BRAF-, EGFR- oder KRAS-Mutationen19
- IDH-Mutationen oder 1p/19q-Kodeletionen bei Gliomen20
- molekulare Subtypen des Mammakarzinoms (z.B. Luminal A/B, HER2+, triple-negativ)21
- DNA-Methylierung-basierte Subklassen von Hirntumoren22
Diese Ansätze beruhen auf der Hypothese, dass molekulare Veränderungen auch zu subtilen morphologischen Veränderungen führen, die zwar für das menschliche Auge schwer erfassbar, für KI-Modelle jedoch durch Trainingsdaten erlernbar sind. So kann etwa eine veränderte Tumorzellkernform, ein spezifisches Wachstumsmuster oder eine bestimmte Zusammensetzung des Stromas Hinweise auf genetische Aberrationen liefern.
Ein wesentlicher Vorteil dieser KI-basierten Verfahren besteht in ihrer Eignung als prädiagnostisches Screeninginstrument: Sie ermöglichen die frühzeitige Identifikation verdächtiger Fälle, bei denen eine weiterführende molekulare Testung indiziert ist, und tragen so zur Beschleunigung des diagnostischen Prozesses und zur Optimierung der Ressourcennutzung bei. Dies ist insbesondere in Settings mit begrenztem Ressourcenbudget oder eingeschränkter Verfügbarkeit molekularer Infrastruktur von hoher Relevanz.
Gleichzeitig ist zu betonen, dass diese bildbasierten Prädiktionen aktuell noch nicht den diagnostischen Goldstandard molekularbiologischer Verfahren ersetzen können. Die Modelle zeigen zwar in validierten Studien teils sehr hohe AUC-Werte (Area Under the Curve, ein Wert für die Genauigkeit der Methode), unterliegen jedoch Limitationen hinsichtlich Generalisierbarkeit, Robustheit bei unterschiedlichen Präparationsarten und Scannerfabrikaten sowie extern validierten Cutoffs für die klinische Entscheidungsfindung.
Transparenz verbessern – Validierung vorantreiben: Aktuelle Entwicklungen zielen darauf ab, die Transparenz (Explainability) dieser Modelle zu verbessern und deren klinische Validierung in multizentrischen Kohorten voranzutreiben. In Zukunft könnten diese bildbasierten molekularen Prädiktionen als Bestandteil eines integrierten KI-gestützten Diagnosesystems dienen, das morphologische, immunhistochemische und molekulare Information zu einem umfassenden Tumorprofil zusammenführt.23
Herausforderungen und ethische Aspekte
Trotz der dynamischen Entwicklung und vielversprechenden Ergebnisse im Bereich der KI-gestützten Pathologie bestehen weiterhin erhebliche Herausforderungen, die einer breiten Implementierung in der klinischen Praxis entgegenstehen. Diese betreffen sowohl technische, konzeptionelle als auch regulatorische Aspekte.
- Mangel an standardisierten Datensätzen und Annotationen:
Ein zentrales Hindernis ist der begrenzte Zugang zu qualitativ hochwertigen, annotierten Bilddatensätzen, die für das Training und die Validierung von KI-Modellen unerlässlich sind. Viele aktuell verfügbare Datensätze stammen aus Einzelzentren, sind nicht repräsentativ für die Diversität der klinischen Realität (z.B. unterschiedliche Fixierung, Färbung, Scannerhersteller) und enthalten insbesondere bei seltenen Tumorentitäten nur unzureichend gelabelte Fälle. Dies schränkt die Generalisierbarkeit und Robustheit der Modelle erheblich ein.
- Algorithm Bias und mangelnde Diversität:
Die Leistungsfähigkeit von Deep-Learning-Modellen ist stark abhängig von der Zusammensetzung der Trainingsdaten. Sind bestimmte klinische oder demografische Gruppen unterrepräsentiert (z.B. Gewebeproben aus nicht-westlichen Ländern, Kinder, bestimmte Ethnien), kann dies zu einem „Algorithm Bias“ führen. Solche Verzerrungen bergen das Risiko diagnostischer Fehlklassifikationen und können bestehende gesundheitliche Ungleichheiten unbeabsichtigt verstärken.
- Begrenzte externe Validierung und fehlende Reproduzierbarkeit:
Viele in der Literatur präsentierten Modelle zeigen hervorragende Ergebnisse bei der internen Validierung, versagen jedoch bei der Anwendung auf externe Kohorten. Die Gründe hierfür liegen u.a. in technischer Heterogenität, Unterschieden im klinischen Workflow sowie fehlender Harmonisierung von Datenformaten. Zudem mangelt es oft an öffentlich zugänglichem Code oder an dokumentierten Trainings- und Validierungsprotokollen, was die Reproduzierbarkeit einschränkt.
- Regulatorische Unsicherheiten und fehlende Zulassungsverfahren:
Im Gegensatz zur klassischen In-vitro-Diagnostik fehlt für KI-basierte diagnostische Systeme derzeit vielfach ein klar definierter regulatorischer Rahmen, insbesondere in Europa unter der neuen IVDR-Verordnung. Fragen nach der Klassifizierung als Medizinprodukt, der Nachweispflicht klinischer Wirksamkeit und der Verantwortlichkeit im Fall diagnostischer Fehlentscheidungen sind bislang unzureichend geklärt. Auch Aspekte wie Modellaktualisierung („continuous learning“) und Softwareversionierung werfen regulatorische Herausforderungen auf.24
- Explainability und Vertrauen in KI-gestützte Entscheidungen:
Ein zentrales Kriterium für die klinische Akzeptanz von KI-Systemen ist deren Erklärbarkeit (Explainability). Klinisch tätige Patholog:innen und Onkolog:innen müssen nachvollziehen können, wie ein KI-System zu einem bestimmten Befund oder einer Klassifikation gelangt ist – insbesondere wenn diese Entscheidungen Auswirkungen auf Therapieentscheidungen haben. Verfahren wie Heatmaps, Attention-Maps oder Layer-wise Relevance Propagation (LRP) bieten erste Ansätze, um Entscheidungswege visualisierbar zu machen.25 Dennoch bleiben viele Modelle aufgrund ihrer Komplexität weitgehend „Black Boxes“, was Vertrauen und Haftungsfragen erschwert.
Perspektiven: Integration in klinische Arbeitsabläufe
Der zukünftige klinische Einsatz von KI in der Pathologie zielt auf eine nahtlose Integration in bestehende diagnostische Workflows, ohne diese zu ersetzen, sondern sie gezielt zu erweitern und zu strukturieren. Dabei geht es nicht allein um Effizienzsteigerung, sondern um die systematische Einbindung KI-gestützter Auswertungen in die interdisziplinäre onkologische Entscheidungsfindung.
Integration in molekulare Tumoboards: Ein zentrales Anwendungsfeld ist die Unterstützung molekularer Tumorboards, in denen zunehmend große Datenmengen (histologische, molekulare, immunologische) in kurzer Zeit konsolidiert und klinisch interpretiert werden müssen. KI-Systeme könnten hier vorab relevante Bildregionen identifizieren, immunologische Parameter quantifizieren oder mutmaßliche molekulare Subtypen vorhersagen – etwa durch Vorschläge zur TMB-Schätzung oder MSI-Detektion. Solche Voranalysen könnten strukturiert in die Tumorboard-Vorlagen integriert und anschließend validiert bzw. überstimmt werden.
Konkrete Anwendung in der Diagnostik: Die klinische Translation KI-gestützter Bildanalysesysteme schreitet dabei nicht nur im Forschungssetting voran, sondern hat bereits konkrete Anwendungen im diagnostischen Alltag gefunden. Mehrere KI-Tools wurden inzwischen von der U.S. Food and Drug Administration (FDA) zugelassen – etwa zur quantitativen Auswertung von Mitoseaktivität, zur PD-L1-Expression oder zur automatisierten Tumorsegmentierung.Diese regulatorischen Freigaben markieren wichtige Meilensteine für den breiteren klinischen Einsatz, da sie die Validierung und Sicherheit der Algorithmen unterstreichen.
Besonders hervorzuheben sind in diesem Kontext Unternehmen wie Indica Labs und Owkin Diagnostics, die kommerzielle, validierte Softwarelösungen für die digitale Pathologie entwickeln. Indica Labs bietet mit seiner Plattform HALO AI ein modulares System zur Bildanalyse, das bereits für zahlreiche Aufgaben – von der Tumorerkennung bis zur Immunzellquantifizierung – in klinischen Studien und Diagnostik verwendet wird. Owkin Diagnostics wiederum fokussiert sich auf KI-basierte Vorhersagemodelle molekularer Tumorprofile und arbeitet eng mit akademischen und industriellen Partnern zusammen, um die Integration dieser Methoden in die Routinepathologie zu ermöglichen.
Auch im deutschsprachigen Raum finden sich erste konkrete Anwendungen dieser Technologien: So nutzen erste Zentren ausgewählte KI-basierte Bildanalysesysteme im Rahmen der täglichen diagnostischen Praxis – insbesondere zur Unterstützung bei immunhistochemischen Auswertungen und zur Qualitätskontrolle. Diese Beispiele belegen, dass KI längst nicht mehr nur ein theoretisches Innovationsversprechen ist, sondern zunehmend Teil realer, zugelassener und validierter Diagnostikprozesse wird.
Automatisierte Befundgenerierung: Parallel dazu schreitet auch die Entwicklung strukturierter Befundsysteme voran, bei denen KI-basierte Analysen direkt in den digitalen Workflow eingebettet werden können. Ein vielversprechender Anwendungsbereich ist die automatisierte Befundgenerierung, bei der quantitative Bildanalysen, standardisierte Terminologien und elektronische Patientenakten miteinander verknüpft werden. Erste Systeme in Form von sog. „reporting assistants“26 sind bereits in Entwicklung und können aus den Ergebnissen KI-gestützter Auswertungen automatisch vorformulierte Textbausteine für den pathologischen Befund erzeugen. Dadurch lassen sich nicht nur Zeitressourcen einsparen, sondern auch Konsistenz und Vergleichbarkeit von Diagnosen verbessern – insbesondere in großen klinischen Netzwerken oder multizentrischen Studien. Ein weiteres Innovationsfeld ist die automatisierte Detektion immunrelevanter Zielstrukturen, wie z.B. PD-L1-Expressionsmuster, TLS (tertiäre lymphoide Strukturen) oder das räumliche Verhältnis von Effektor-T-Zellen zum Tumorparenchym. Diese Analysen könnten ein personalisiertes Immunprofiling ermöglichen – standardisiert, objektiv und reproduzierbar.
Biomarker-Identifizierung: Langfristig bietet KI zudem die Möglichkeit, neue Biomarker zu identifizieren, indem sie hochdimensionale Daten aus Bildgebung, Genomik und Transkriptomik miteinander verknüpft. Solche Modelle könnten latente Muster aufdecken, die für das menschliche Auge nicht unmittelbar erkennbar sind, aber signifikant mit klinischen Endpunkten korrelieren – etwa überlebensrelevante Immunmuster oder Therapieansprechprofile.
„Digitaler Immunzwilling“: Ein besonders zukunftsweisendes Konzept ist der „digitale Immunzwilling“27: eine computergestützte Modellierung individueller Immunantworten auf Basis histopathologischer, molekularer und klinischer Daten. Ziel ist, patientenspezifisch vorhersagen zu können, wie das Immunsystem auf eine bestimmte Tumorentität oder Therapie (z.B. Checkpoint-Inhibitoren) reagiert – ein Schritt in Richtung simulationsgestützte, personalisierte Onkologie.
Fazit
Künstliche Intelligenz steht kurz davor, ein integraler Bestandteil der modernen Pathologie zu werden. Insbesondere in der Immunpathologie bietet sie neue diagnostische und prognostische Möglichkeiten – von der objektiven Quantifizierung immunologischer Zellpopulationen über die Prädiktion molekularer Marker bis hin zur Identifikation bislang unbekannter Muster im Tumormikroenvironment.
KI wird dabei nicht als Ersatz, sondern als präzise und skalierbare Ergänzung zur pathologischen Expertise verstanden. Ihr Einsatz kann die Standardisierung verbessern, Arbeitsabläufe optimieren und die Grundlage für datengetriebene, personalisierte Therapieentscheidungen erweitern.
Damit das volle Potenzial dieser Technologien verantwortungsvoll ausgeschöpft werden kann, bedarf es belastbarer Validierungsstudien, interoperabler Datenstandards, transparenter Entscheidungslogiken und klarer regulatorischer Leitlinien – mit dem Ziel, Vertrauen, Sicherheit und klinischen Nutzen nachhaltig zu gewährleisten.
| FACT-BOX |
|---|
|

























































































