Systembiologie in der chronischen Niereninsuffizienz
Systembiologie
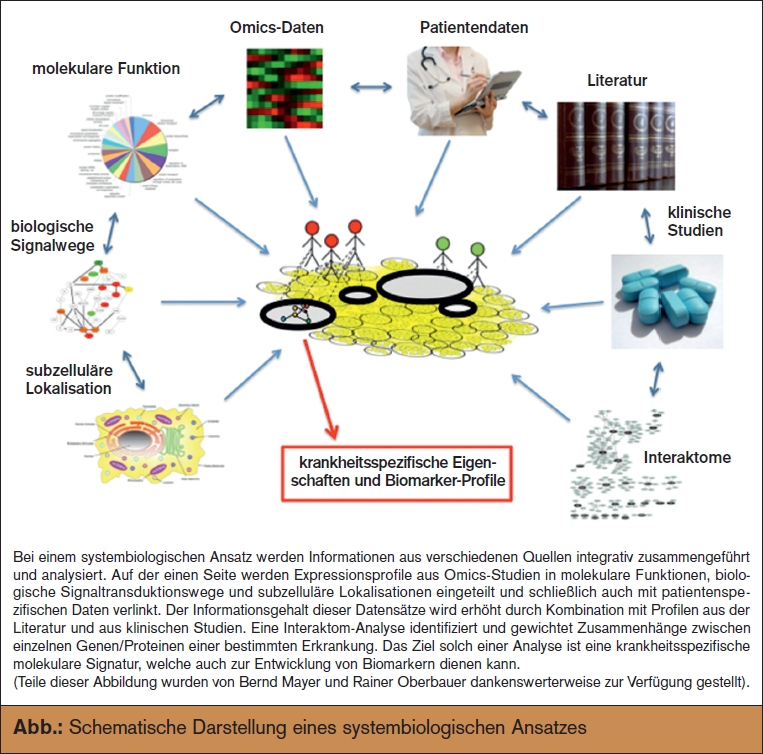 Obwohl die Systembiologie keine ganz junge Wissenschaft ist, kam es erst in den letzten zwei Jahrzehnten zu wesentlichen Veränderungen in der systemiologischen biomedizinischen Forschung, die eine komplexe, integrative Datenanalyse und Interpretation ermöglicht haben. Es wurden experimentelle Analysen entwickelt, die die simultane Erfassung von Veränderungen einer Vielzahl an Molekülen eines zellulären Systems ermöglicht haben. Diese Methoden werden für gewöhnlich unter dem Begriff „Omics“ zusammengefasst, angelehnt an den ersten in diesem Zusammenhang verwendeten Begriff „Genomics“, bei dem die Genexpression (RNA) des gesamten Genoms gleichzeitig erfasst wird. Auch bei microRNAs (miRNomics), Proteinen (Proteomics) und Metaboliten (Metabolomics) ermöglichen neue experimentelle Methoden die umfassende Expressionsanalyse vieler dieser Moleküle aus nahezu jeder Art von Gewebe oder Körperflüssigkeit (> Abb.). Die Verbindung leistungsfähiger Messmethoden mit computergestützten Datenerfassungssystemen und mit potenter Analysesoftware ermöglichte zudem eine sinnvolle Analyse und Interpretation der Ergebnisse der Omics- Studien. Zur Veranschaulichung: Ein einzelnes Genomics-Experiment von einer Nierenbiopsie liefert derzeit Informationen zu Expressionswerten von ca. 30.000 Genen. Analysiert man 50 verschie dene Patientenproben, so erhält man eine Datenmatrix mit 50 x 30.000 = 1.5 Mio. Datenpunkten. Diese immensen Datenmengen können nur mithilfe verschiedener, größtenteils öffentlich zugänglicher integrativer Datenbanken und Softwarelösungen analysiert oder mit anderen publizierten Daten in Verbindung gebracht werden. Zu diesen bioinformatischen Datenquellen zählen KEGG (Kyoto Encyclopedia of Genes and Genomes), PANTHER (Protein ANalysis THrough Evolutionary Relationships), DAVID (Database for Annotation, Visualization and Integrated Discovery), GEO (Gene Expression Omnibus), Gene Ontology und andere. Die in diesen Datenbanken gespeicherten Informationen über die biologischen Funktionen eines bestimmten Moleküls X bzw. über molekularen Prozesse, an denen das Molekül teilnimmt, werden dann mithilfe geeigneter Applikationen im Sinne einer Pathway-Analyse abgerufen. Werden zudem noch klinische Daten der Patienten in diese Analyse integriert, so wird u. U. eine neue molekulare Unterteilung vorher homogen erscheinender Krankheiten ermöglicht, deren Informationsgehalt über die traditionellen Risikofaktoren und Krankheitsmarker (klinisch, labordiagnostisch, histologisch) hinausgeht.
Obwohl die Systembiologie keine ganz junge Wissenschaft ist, kam es erst in den letzten zwei Jahrzehnten zu wesentlichen Veränderungen in der systemiologischen biomedizinischen Forschung, die eine komplexe, integrative Datenanalyse und Interpretation ermöglicht haben. Es wurden experimentelle Analysen entwickelt, die die simultane Erfassung von Veränderungen einer Vielzahl an Molekülen eines zellulären Systems ermöglicht haben. Diese Methoden werden für gewöhnlich unter dem Begriff „Omics“ zusammengefasst, angelehnt an den ersten in diesem Zusammenhang verwendeten Begriff „Genomics“, bei dem die Genexpression (RNA) des gesamten Genoms gleichzeitig erfasst wird. Auch bei microRNAs (miRNomics), Proteinen (Proteomics) und Metaboliten (Metabolomics) ermöglichen neue experimentelle Methoden die umfassende Expressionsanalyse vieler dieser Moleküle aus nahezu jeder Art von Gewebe oder Körperflüssigkeit (> Abb.). Die Verbindung leistungsfähiger Messmethoden mit computergestützten Datenerfassungssystemen und mit potenter Analysesoftware ermöglichte zudem eine sinnvolle Analyse und Interpretation der Ergebnisse der Omics- Studien. Zur Veranschaulichung: Ein einzelnes Genomics-Experiment von einer Nierenbiopsie liefert derzeit Informationen zu Expressionswerten von ca. 30.000 Genen. Analysiert man 50 verschie dene Patientenproben, so erhält man eine Datenmatrix mit 50 x 30.000 = 1.5 Mio. Datenpunkten. Diese immensen Datenmengen können nur mithilfe verschiedener, größtenteils öffentlich zugänglicher integrativer Datenbanken und Softwarelösungen analysiert oder mit anderen publizierten Daten in Verbindung gebracht werden. Zu diesen bioinformatischen Datenquellen zählen KEGG (Kyoto Encyclopedia of Genes and Genomes), PANTHER (Protein ANalysis THrough Evolutionary Relationships), DAVID (Database for Annotation, Visualization and Integrated Discovery), GEO (Gene Expression Omnibus), Gene Ontology und andere. Die in diesen Datenbanken gespeicherten Informationen über die biologischen Funktionen eines bestimmten Moleküls X bzw. über molekularen Prozesse, an denen das Molekül teilnimmt, werden dann mithilfe geeigneter Applikationen im Sinne einer Pathway-Analyse abgerufen. Werden zudem noch klinische Daten der Patienten in diese Analyse integriert, so wird u. U. eine neue molekulare Unterteilung vorher homogen erscheinender Krankheiten ermöglicht, deren Informationsgehalt über die traditionellen Risikofaktoren und Krankheitsmarker (klinisch, labordiagnostisch, histologisch) hinausgeht.
Chronische Niereninsuffizienz
Obwohl vermutlich ca. 10–13 % der Gesamtbevölkerung an einer chronischen Niereninsuffizienz leiden, sind die diagnostischen als auch therapeutischen Möglichkeiten trotz intensiver Forschung und vielen neuen Erkenntnissen weiterhin limitiert. Die Diagnose einer Nierenerkrankung basiert nach wie vor u. a. auf der Harndiagnostik (Proteinurie, Albuminurie, Hämaturie, Auffälligkeiten im Sediment) und in ausgewählten Fällen auf den pathohistologischen Veränderungen in der Nierenbiopsie. Obwohl verschiedene Biomarker zur Progressionsabschätzung der chronischen Niereninsuffizienz publiziert wurden, die in der Zukunft eine klinisch relevante Rolle spielen könnten, gelten zurzeit konservative Parameter, wie z. B. Serum-Kreatinin (oder die glomeruläre Filtrationsrate), Proteinurie/Albuminurie, Cystatin C oder das klinische Ansprechen auf eine RAAS-Blockade weiterhin als die besten Marker, um die renale Prognose eines individuellen Patienten abschätzen zu können. Zusätzlich haben sich die Eckpfeiler der Therapie der meisten nephrologischen Erkrankungen (RAAS-Blockade, konsequente antihypertensive Therapie, Immunsuppression) in den letzten Dekaden nicht wirklich verändert. Hier liefern systembiologische Analysen vor allem von Omics-Studien aus Nierengewebe interessante Ergebnisse, die sowohl neue diagnostische Möglichkeiten eröffnen als auch neue und teilweise unterwartete Einblicke in die Pathophysiologie chronischer Nierenerkrankungen liefern. Petersen et al. (J Clin Invest 2004) untersuchten mikrodissezierte Glomerula bei Patienten mit Lupus-Nephritis WHO-Klasse III und IV. Obwohl die klinischen Parameter betreffend Nierenfunktion, Proteinurie, Hämaturie und Histologie durchaus vergleichbar waren, zeigten sich in den mRNA-Profilen starke intraindividuelle Unterschiede.
Es konnten 4 verschiedene molekulare Signaturen identifiziert werden, nämlich:
- Interferon-alpha-Response-Elemente
- ein myelomonozytäres Profil
- extrazelluläre Matrix- und Fibrose-Gene und
- ein B-Lymphozytengencluster
Unterschiede in der Genexpression als auch in der Aktivierung bestimmter intrazellulärer Pathways konnten ebenso in Glomerula von Patienten mit fokal-segmentaler Glomerulosklerose (FSGS) gezeigt werden (Hodgin et al., Am J Pathol 2010). Bei Patienten mit bioptisch gesicherter diabetischer Nephropathie konnten Schmid und Mitarbeiter (Diabetes 2006) zeigen, dass sich die Genexpressionsmuster des tubulointerstitiellen Kompartments bei Patienten mit stabilem Verlauf und mit einer progressiven Verschlechterung der Nierenfunktion signifikant unterschieden. So zeigten nur progrediente, nicht jedoch stabile Patienten eine deutliche Aktivierung von inflammationsabhängigen Genen, die mit einem spezifischen NF-kappa-B-Modul assoziiert waren. Durch die Kombination von globaler Genexpression mit leistungsfähigen bioinformatischen Analysemethoden konnten Berthier et al. (Diabetes 2009) die Aktivierung des Jak- Stat-Pathways bei progressiver diabetischer Nephropathie identifizieren. Unsere Arbeitsgruppe (Rudnicki et al., Lab Invest 2009) konnte schließlich die Aktivierung der Hypoxie- Response- und VEGF-Pathways in proximalen Tubuluszellen bei Patienten mit progressiven proteinurischen Nierenerkrankungen zeigen, wobei VEGF als wichtiges Zielgen dieser intrazellulären Signaltransduktionskaskaden bei progressiven Nierenerkrankungen vermindert exprimiert wurde.
Biomarker
Omics-Methoden und systembiologische Analysen bieten ein vielversprechendes Potenzial zur Identifikation neuer krankheits- und gewebespezifischer Biomarker. Ein schönes Beispiel für eine nicht-supervidierte Analyse, bei der eine an sich homogene histologische Kohorte durch die Genexpressionsanalyse in mehrere Gruppen geteilt wird, wurde von Henger und Kollegen (Kidney Int 2004) publiziert. In dieser Arbeit konnte anhand der Genexpression das tubulointerstitielle Kompartment von Nierenbiopsien in eine eher inflammierte und in eine eher fibrosierte Kohorte geteilt werden. Zusätzlich wurden vor allem in der inflammatorischen Gruppe 7 Gene identifiziert, die eine signifikante Korrelation mit dem klinischen postbioptischen Verlauf zeigten. In unserer Arbeit aus dem Jahr 2009 konnten wir mit Hilfe von Genomics- Methoden zeigen, dass die Expression von VEGF und HIF-1_ einen besseren prädiktiven Wert für den postbioptischen Abfall der GFR hatte als konventionelle Faktoren wie der Grad der histologischen Schädigung, das Ausmaß der Proteinurie oder die Nierenfunktion bei der Biopsie. In einer rezenten Arbeit konnten wir zudem 3 weitere Markermoleküle TRIB1, BMP7 und THBS1 identifizieren, die ebenso eine signifikant bessere Vorhersagekraft für die renale Prognose hatten als die herkömmlichen Marker (Neuwirt et al., Submitted). Ebenso gibt es auf dem Gebiet der Proteomics, Metabolomics und miRNomics neue und aufregende systembiologische Ansätze in der Biomarkerforschung, deren ausführliche Darstellung jedoch den Rahmen dieses Artikels sprengen würde.
FACT BOX
- Systembiologie und Omics-Methoden versuchen ein gegebenes biologisches System in seinem gesamten Ist-Zustand zu erfassen und Hypothesen zu generieren.
- Bei chronischer Niereninsuffizienz wurden durch Genomics-Studien Signaltransduktionswege im Nierengewebe identifiziert, die mit dem Schweregrad der Erkrankung oder dem klinischen Verlauf korrelieren.
- Durch Omics-Studien und systembiologische Methoden wurden verschiedene Biomarkerkandidaten identifiziert, die in prospektiven Untersuchungen in verschiedenen Kohorten validiert werden müssen.