Biomarker zur minimal invasiven und frühen Diagnostik von Tumorerkrankungen – Reicht ein Blutstropfen?
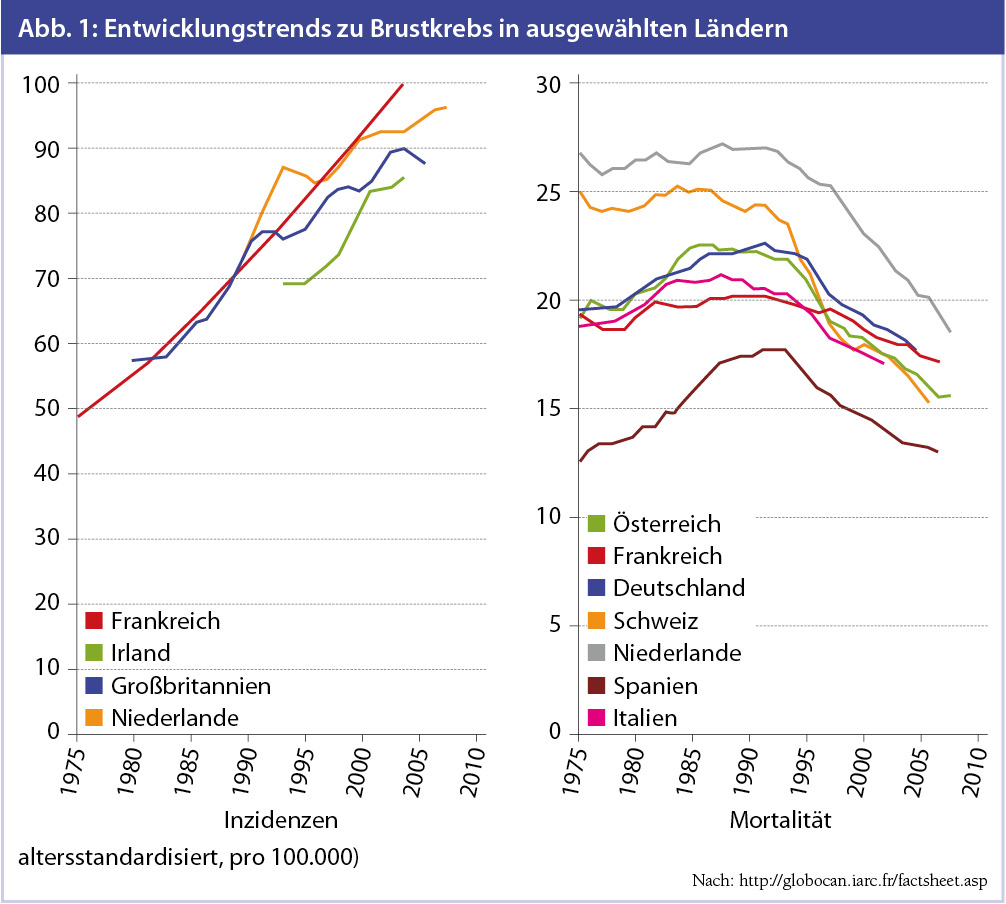
Sofern die Entwicklungen und der Trend des Anstiegs von Krebserkrankungen der letzten Jahrzehnte anhalten, wird es zu einem Anstieg von 12,7 Mio. Krebsneuerkrankungen im Jahr 2008 auf 22,2 Mio. im Jahr 2030 kommen, wird in einer rezenten Arbeit geschätzt (Bray et al., 2012). Für Brustkrebs sind diese Trends ebenso zu verzeichnen (Abb. 1). Obwohl Brustkrebs in hoch entwickelten Ländern die häufigste Krebserkrankung ist, sind aufgrund der verbesserten Therapien und Vorsorgeprogramme die Mortalitäten bei Brustkrebs im Sinken begriffen. Dennoch ist Brustkrebs bei Frauen die häufigste krebsabhängige Todesursache in Entwicklungs- und hochentwickelten Ländern.
Krebs-Frühdiagnostik durch Biomarker
Die Krebsfrüherkennung, d. h. Diagnose von frühen Krankheitsstadien, kann insbesondere die Heilungschance steigern. Im Fall von Brustkrebs leisten dazu die Mammografie sowie MRI und Brustultraschall einen besonders wertvollen Dienst, wenngleich diese bildgebenden Verfahren auch ihre Limitationen aufweisen. Um die Detektion sehr früher Tumorstadien weiter zu verbessern, werden derzeit molekulare Techniken eingesetzt, um Biomarker zu identifizieren, welche die Früherkennung im Blut, Serum, Stuhl, Urin, Sputum oder Speichel von Probanden ermöglichen sollen. Dazu eignen sich tumorspezifische Nukleinsäure-Marker, wie etwa DNA-Methylierung und miRNA, und auch tumorspezifische Proteine und Metaboliten als potenzielle Tumormarker.
Zur Serumdiagnostik von Darmkrebs und auch von Lungenkrebs sind sei 2011 DNA-Methylierungs-basierte PCR-Tests am Markt, welche mit 3,5 ml Blutplasma arbeiten (Kneip et al., 2011; Warren et al., 2011). Derzeit sind diese Tests primär zur Absicherung von fraglichen Befunden geeignet, aber aufgrund der relativ hohen Kosten und des Arbeitsaufwandes weniger für ein breites Screening-Verfahren geeignet. Sofern die Probenlogistik und PCR-Analytik im Hochdurchsatz etabliert wäre, wie dies beispielsweise zur Virustestung von Spender-Blut seit vielen Jahren der Fall ist, sind solche Nukleinsäure-basierte Tests durchaus für ein Screening geeignet.
Tumor-Autoantikörper in Serum von Patienten stellen eine weitere Option dar, welche gegenüber den genannten Biomolekülen als Biomarker zum Einsatz für ein breites Screening besonders geeignet sind, da diese relativ einfach mittels ELISA messbar sind.
Projekt zur Serumdiagnostik mit Tumor-Autoantikörpern
Tumor-Autoantikörper im Serum der Patienten werden vom Immunsystem gegen tumorspezifische, von Krebszellen aberrant exprimierte Proteine gebildet und sind ein früher Hinweis auf (Brust-)Krebs (Luna Coronell et al., 2012). Um diese krankheitsspezifischen Tumor-Autoantikörper-Muster zu identifizieren, können verschiedene Verfahren eingesetzt werden.
Protein-Mikroarray-basiertes Biomarker-Screening: Wir haben dazu gemeinsam mit der Abteilung Spezielle Gynäkologie (Prof. Christian Singer, Medizinische Universität & AKH Wien) und dem Institut für angewandte Mikrobiologie (Prof. Florian Rüker, Department für Biotechnologie, Universität für Bodenkultur, Wien), ein durch den WWTF gefördertes Projekt gestartet, in welchem bereits etablierte Verfahren kombiniert werden, um eine möglichst hohe Sensitivität und Spezifität zur Detektion von Brustkrebs gegenüber gutartigen (benignen) Knoten und gesunden Probandinnen zu erzielen.
Dabei werden in Vorarbeiten identifizierte seroreaktive Expressionsklone, welche Brustkrebs-spezifische Gene exprimieren und mit Immunglobulin von Patienten reaktiv sind, zusammen mit rekombinant exprimierten Humanproteinen aus 15286-cDNA-Klonen einer humanen Klonsammlung, für die Herstellung von Protein-Microarrays verwendet.
Immunglobulin-Profile zur Unterscheidung maligner, benigner und gesunder Probanden: Mit etwa 30 Mikroliter Serum von Patienten mit Brustkrebs, benignen Knoten in der Brust und gesunden Kontroll-Serumproben konnte somit ein Screening durchgeführt werden, um bestmöglich zwischen den 3 Klassen von malignen, benignen und gesunden Serumproben zu unterscheiden.
Diese Immunglobulin-Profile werden zurzeit mittels bioinformatischer Analyse zur Unterscheidung der 3 Klassen definiert. In ersten Analysen konnten durch die Kombination von etwa 50–100 antigenischen Proteinen vielversprechende Klassifikationserfolge erzielt werden. Damit wurden in einer „ROC-Analyse“ AUC-Werte von 0,84–0,98 erhalten (Abb. 2).
Phage-Display-Technologie: Zusätzlich zu den Protein-Mikroarray-basierten Biomarker-Screenings setzten wir die Phage-Display-Technologie ein, um seroreaktive antigenische Peptide welche ebenso, wie die hochmolekularen Proteine, zur Detektion von Tumor-Autoantikörpern eingesetzt werden können. Dazu werden mit Immunglobulinen (IgG) aus Serum-Pools von Patienten und Kontrollen die für die 3 Probanden-Gruppen (malign, benign, gesund) spezifischen Phagen-Populationen angereichert, welche durch zufällige Gensequenzen definierte Peptide an ihren Pili exprimieren. Diese für jeweils eine Probanden-Gruppe spezifisch selektierte Phagenpopulation wird mittels hochparalleler DNA-Sequenzierung analysiert und somit die zugehörigen Peptide definiert. In einem ersten Experiment konnten wir damit von 75.000 Sequenzen über 3.000 probandengruppenspezifische Peptide definieren. Davon werden wiederum mittels Peptid-Microarray-Analyse von Serumproben die Peptid-Klassifikatoren bestimmt, welche eine bestmögliche Unterscheidung zwischen den Probandinnen mit malignen und benignen Knoten sowie gesunden Kontrollprobandinnen ermöglicht.
Validierung: Die Tumorkandidatenmarker-Panels aus den oben erwähnten Proteinen und den mittels Phage-Display identifizierten Peptiden, werden im klinischen Forschungslabor mit dem Test- und Validierungskollektiv validiert. Zudem werden alle über den Projektzeitraum gesammelten Proben analysiert (n = 1.200; retrospektive Studie).
Letztendlich wird mit diesem Projekt ein Prototyp-Test generiert, der für die Validierung mit einigen hundert Proben geeignet ist. Nachfolgende Validierungsschritte können mit longitudinalen retrospektiven Serumproben oder aber müssen in einem prospektiven Studiensetting durchgeführt werden.
Indikationsspezifische Antigenpanels für Darm-, Prostata-, Lungen- und Brustkrebs: In 2 ähnlich gelagerten Projekten haben wir inzwischen den am AIT hergestellten 16-k-Proteinchip zur Identifikation von Darmkrebs und Prostatakrebs (in Kooperation mit Prim. Univ.-Doz. Friedrich Längle und Prim. Univ.-Doz. Johann Hofbauer, LKH Wiener Neustadt – gefördert durch die NFB [Niederösterreichische Forschungs- und Bildungsges.m.b.H]) und Lungenkrebs (in Kooperation mit Prof. Andrea Gsur, Institut für Krebsforschung, MUW – gefördert durch den Jubiläumsfonds der Österreichischen Nationalbank) durchgeführt. Für diese Indikationen konnten im Screening ebenso gute Ergebnisse erzielt werden. Ein Vergleich der für die verschiedenen 4 großen Krebserkrankungen identifizierten Panels an Tumormarker-Kandidaten zeigte kaum Überlappung dieser „Antigen-Panels“. Somit kann davon ausgegangen werden, dass die definierten Marker indikationsspezifisch sind.
Wie schon dargestellt, müssen nun diese Kandidatenmarker eingehend validiert werden. Dies erfolgt in einem ersten Schritt mit einem unabhängigen Serumprobenkollektiv unter Verwendung von Protein-Microarrays und „Bead-Arrays“. Dazu sind wir immer auf die Verfügbarkeit von klinischen Proben angewiesen und wollen hier auch den klinischen Partnern für ihre Unterstützung und den Patienten für ihre Einwilligung zur Verwendung von Probenmaterial für diese Forschungen danken.
AUSBLICK: Obwohl in den vergangenen zwei Jahrzehnten viele Anstrengungen unternommen wurden, um die frühe Diagnostik zu verbessern, sind international die Erfolge sehr beschränkt (Wagner und Srivastava, 2012). Dies hat viele Gründe, ein wohl sehr gewichtiger ist die Verfügbarkeit klinischer Proben – insbesondere die relativ große Anzahl an benötigten Probanden zur Validierung von Kandidatenmarkern. Hierzu hat ebenso die beschriebene Tumor-Autoantikörper-basierte Analytik den Vorteil, dass diese Analysen mit wenigen Mikrolitern von den meist sehr begrenzt verfügbaren Probenkollektiven erfolgreich durchgeführt werden können. Mit den derzeit verfügbaren technischen Möglichkeiten können mit geringen Mengen an Probenmaterial bereits sehr eindrucksvolle Forschungsergebnisse erzielt werden. Der Weg hin zu validierten Biomarkern und Tests zur Verbesserung der minimal invasiven Diagnostik kann nur in Kooperation verschiedener Disziplinen und vor allem durch das Engagement von Klinikern zur systematischen Sammlung von Patientenproben gelingen.
Technisch gesehen „reicht ein Blutstropfen“ – allerdings fehlen derzeit die validierten Biomarker.