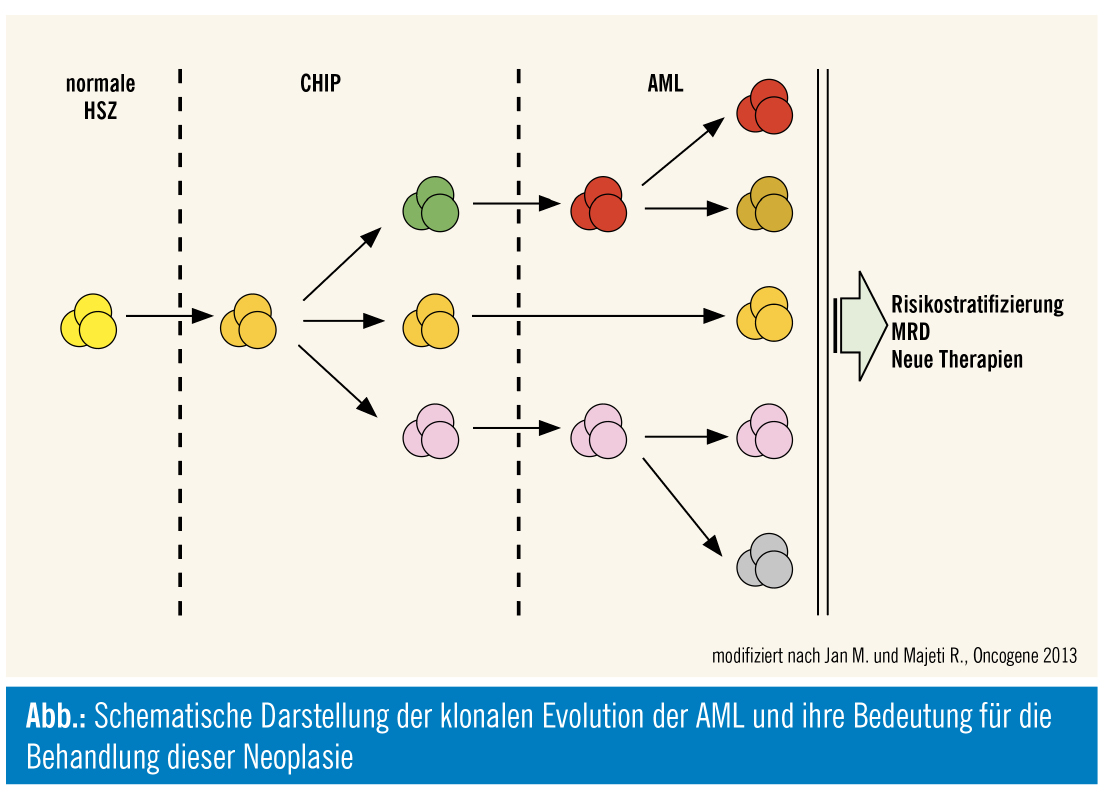
Bedeutung der klonalen Evolution für die Behandlung hämatologischer Neoplasien
Die kontinuierliche Anhäufung von genetischen und epigenetischen Aberrationen in hämatopoetischen Stammzellen (HSZ) kann diese in präleukämische HSZ bzw. leukämische Stammzellen (LSZ) transformieren und damit die Entstehung einer akuten myeloischen Leukämie (AML) verursachen. Dieses Modell der klonalen Evolution ist jedoch nicht nur für die Entstehung der AML wichtig, sondern ist auch für deren Behandlung von Bedeutung. Dies soll im vorliegenden Artikel genauer dargestellt werden.
Klonale Evolution der AML
Die AML wird durch genetische und epigenetische Aberrationen in HSZ verursacht1. Tatsächlich können solche Veränderungen bei fast allen PatientInnen mit AML detektiert werden2. Darüber hinaus konnte das Auftreten von Leukämie-assoziierten Mutationen auch bei einem kleinen Teil von gesunden ProbandInnen nachgewiesen werden3, 4. Dies deutet auf eine klonale Hämatopoese bei den betroffenen Personen hin und wird als klonale Hämatopoese von unbestimmtem Potential (CHIP) bezeichnet5. Obwohl CHIP-Mutationen in vielen unterschiedlichen Genen beschrieben wurden, sind dabei häufig Regulatoren der Epigenetik, wie z. B. DNMT3A oder TET2, betroffen. Die Häufigkeit von CHIP nimmt mit dem Alter zu und ist mit einem erhöhten Risiko für hämatologische Neoplasien vergesellschaftet. Tatsächlich konnte in funktionellen Studien gezeigt werden, dass CHIP-Mutationen in den HSZ zwar noch keine volle Leukämie auslösen, aber trotzdem einige pathologische Stammzellfunktionen verursachen können (insbesondere eine erhöhte Selbsterneuerungskapazität)5. Aus diesem Grund werden die betroffenen Stammzellen auch als präleukämische HSZ bezeichnet. Da die Lebensspanne dieser präleukämischen HSZ pathologisch verlängert ist, sind sie für das Auftreten von weiteren (epi-)genetischen Aberrationen anfällig. Wenn dadurch zusätzlich die Differenzierung und das Wachstum der Zellen beeinträchtigt werden, kann die präleukämische HSZ in eine LSZ transformieren und die Entstehung einer AML induzieren. Solche sekundären Veränderungen betreffen häufig Gene der intrazellulären Signaltransduktion, wie z. B. RAS und FLT3, aber auch das NPM1-Gen5. Dieser Prozess der Leukämieentstehung wird als klonale Evolution der AML bezeichnet. Bei klonaler Evolution handelt es sich um keinen streng linearen Prozess. Der präleukämische Klon kann sich in mehrere parallel existierende Subklone mit unterschiedlichen Mustern an Aberrationen auftrennen, wovon einzelne, aber auch mehrere in eine LSZ transformieren können5, 6. Auch nach der Entstehung einer AML unterliegen sowohl präleukämische HSZ als auch LSZ der ständigen Evolution. Hier ist die weitere Auftrennung in unterschiedliche Subklone mit spezifischen Mutationsmustern häufig. Aus diesem Grund weist die AML nicht nur eine große Heterogenität zwischen unterschiedlichen PatientInnen auf, auch innerhalb einzelner PatientInnen präsentiert sich die AML als extrem vielschichtig und besteht aus mehreren Haupt- und Subklonen. Auch unterschiedlichste präleukämische HSZ und LSZ koexistieren in jeder AML. Aus dieser genetischen Diversität ergeben sich einige wichtige Aspekte für die Behandlung.
Bedeutung der klonalen Evolution für die Risikostratifizierung der AML: Die molekulare Information, insbesondere das Vorkommen von Mutationen, ist aus den heute gängigen Risikostratifizierungstools der AML nicht mehr wegzudenken. So hat sich gezeigt, dass Mutationen in NPM1 oder CEBPA (biallelisch) mit einer guten Prognose vergesellschaftet sind, während interne Tandemduplikationen in FLT3 (FLT3-ITD) sowie Mutationen in RUNX1, ASXL1 oder TP53 mit einem schlechten Outcome assoziiert sind7. Dies wurde bereits in die aktuelle European LeukemiaNet Risikostratifizierung (ELN 2017) aufgenommen und ist damit Teil des klinischen Alltags. Weniger ist bisher über die Wertigkeit der Mutationslast bekannt. Ältere Sequenzierstudien behandelten Mutationen als dichotome Variablen und berücksichtigten nicht, ob Mutationen in dominanten Hauptklonen der AML zu finden sind (große Mutationslast) oder nur Subklone betreffen (kleine Mutationslast). Über die heute gängigen next-generation sequenc-ing (NGS)-Verfahren steht uns diese Information jedoch über die Bestimmung der Variantenallelfrequenz (VAF) zur Verfügung. Dass die Mutationslast auch für die Risikostratifizierung relevant sein kann, zeigt sich am Beispiel der FLT3-ITD. Nur Fälle mit einer Ratio mutiertes Allel/unmutiertes Allel von ≥ 0,5 weisen tatsächlich eine schlechte Prognose auf und werden von den ELN-Guidelines als schlechter prognostischer Marker akzeptiert7. Daten unserer eigenen Gruppe zeigen jedoch nun, dass die Wertigkeit der Mutationslast offensichtlich für unterschiedliche Gene variieren kann. Bei der Bestimmung von TP53-Mutationen in mehr als 1.500 AML-PatientInnen konnten wir zeigen, dass nicht nur klonale TP53-Mutationen mit schlechter Prognose vergesellschaftet waren, sondern auch subklonale mit kleiner Mutationslast (definiert als VAF < 20 %)8. Diese Daten zeigen, dass sich zukünftige Forschungsprojekte noch vermehrt mit der Auswirkung der Mutationslast und der Zugehörigkeit zu Haupt- oder Subklonen beschäftigen werden müssen.
Bedeutung der klonalen Evolution für die Remissionskontrolle der AML: In der Remissionskontrolle der AML gewinnt die Bestimmung einer minimalen Resterkrankung (MRD) zunehmend an Bedeutung. Hier zeigte sich, dass eine persistierende MRD mit einem hohen Rezidivrisiko assoziiert ist, so dass in diesem Fall alternative oder ergänzende Therapiestrategien diskutiert werden sollten7. In der klinischen Routine werden dafür v. a. die Durchflusszytometrie sowie PCR-basierte Methoden verwendet7, 9. Letztere sind dabei v. a. auf die RUNX1/RUNX1T1-, CBFB/MYH11- und PML/RARA-Translokationen sowie auf NPM1-Mutationen limitiert. Durch den Einsatz von NGS-basierten Methoden kann aber grundsätzlich jede bei Diagnose der AML detektierte Mutation zur MRD-Bestimmung herangezogen werden. Tatsächlich konnten andere Gruppen und wir zeigen, dass die fehlende Clearance von bestimmten Mutationen mit einem erhöhten Rezidivrisiko einhergeht10, 11. Interessant war hier jedoch, dass dies nicht auf alle Mutationen zutrifft, und dass bestimmte Mutationen, wie z. B. DNMT3A oder TET2, trotz fehlender Clearance kein Rezidiv vorhersagen können. Unter dem Gesichtspunkt der klonalen Evolution scheinen diese Aberrationen eher der präleukämischen Phase zuordenbar zu sein. Ihre Nachweisbarkeit dürfte damit eher Ausdruck einer nach wie vor bestehenden CHIP als einer MRD der AML sein. Ob diese Aberrationen für den längerfristigen Nachsorgeverlauf oder für das Auftreten von Therapie-assoziierten Leukämien oder Zweitmalignomen von Bedeutung sind, ist bisher nicht ausreichend geklärt und bedarf weiterer Forschung.
Bedeutung der klonalen Evolution für die Chemoresistenz und das Rezidiv der AML: 60–80 % der jüngeren AML-PatientInnen erreichen mit intensiven Induktionsregimen eine komplette Remission7. Leider entwickelt jedoch ein nicht unbeträchtlicher Teil ein chemoresistentes Rezidiv und verstirbt schlussendlich daran1. Auch hierbei spielt das Konzept der klonalen Evolution eine große Rolle. NGS-Studien konnten zeigen, dass sich das molekulare Profil der AML im zeitlichen Verlauf ändert und sich zum Zeitpunkt des Rezidivs häufig stark vom Diagnosezeitpunkt unterscheidet12. Auch wenn es hier noch kein klares Muster gibt, so zeigte sich, dass dominante Mutationen zum Relaps-Zeitpunkt häufig Chemoresistenz verursachen können und schon als subklonale Veränderung zum Diagnosezeitpunkt nachweisbar waren12, 13. Dies deutet darauf hin, dass die initiale Chemotherapie zwar die bei Diagnose vorliegenden dominanten Klone effektiv beseitigt, dass jedoch Subklone (als Ausdruck eines chemorefraktären LSZ-Kompartiments) bestehen bleiben und im weiteren Verlauf Ausgangspunkt eines therapieresistenten Rezidivs sind. Es ist anzunehmen, dass auch diese Zellkompartimente einer weiteren Chemotherapie-induzierten Evolution und Selektion unterliegen, dies ist jedoch bisher nicht ausreichend untersucht. Diese Modelle der Resistenzentstehung sind auch für die Entwicklung von neuen Therapiestrategien von Bedeutung, da neue Substanzen die Elimination der chemoresistenten LSZ ermöglichen müssen, um lange anhaltende Remissionen zu erzielen. Während dies für einige Substanzen, wie z. B. Venetoclax, der Fall zu sein scheint14, braucht es hier sicher noch mehr Forschung und Entwicklung.
Resüme
AML entsteht durch (epi-)genetische Aberrationen und klonale Evolution in hämatopoetischen Stammzellen. Dies führt zur gleichzeitigen Existenz vieler molekular unterschiedlicher Haupt- und Subklone bei PatientInnen mit dieser Erkrankung. Das kann Einfluss auf wesentliche Punkte der AML-Behandlung, wie z. B. die Risikostratifizierung oder die MRD-Messung, haben. Schlussendlich ist es für die Entwicklung neuer Therapien wichtig, da diese das Konzept der klonalen Evolution berücksichtigen müssen und idealerweise das LSZ-Kompartiment angreifen sollten.