Studienpräsentation: miRNA-Profile unterscheiden Art der Abstoßung und Schädigung in Nierentransplantaten
Unpublizierte laufende Studie
In einer rezenten Studie konnten wir miRNA-Signaturen bei akutem Nierentransplantatversagen („delayed graft function“) und akuter Abstoßung in humanen Spendernieren nachweisen. Insgesamt wurde von 65 Nierenbiopsaten ein genomweites miRNA-Profil erstellt (GeneChip® miRNA Array von Affymetrix). Die 65 Biopsate beinhalteten 30 akute zelluläre Abstoßungen (15 BANFF1, 15 BANFF2), 11 humorale Abstoßungen (C4d-positiv), 14 „delayed graft functions“ (DGF, diagnostizierte akute tubuläre Nekrose) und 10 Protokollbiopsien ohne ein Zeichen einer Schädigung (Kontrollgruppe). Die signifikanten differenzial regulierten miRNA wurden mittels t-Test und Bonferroni-Korrektur ermittelt.
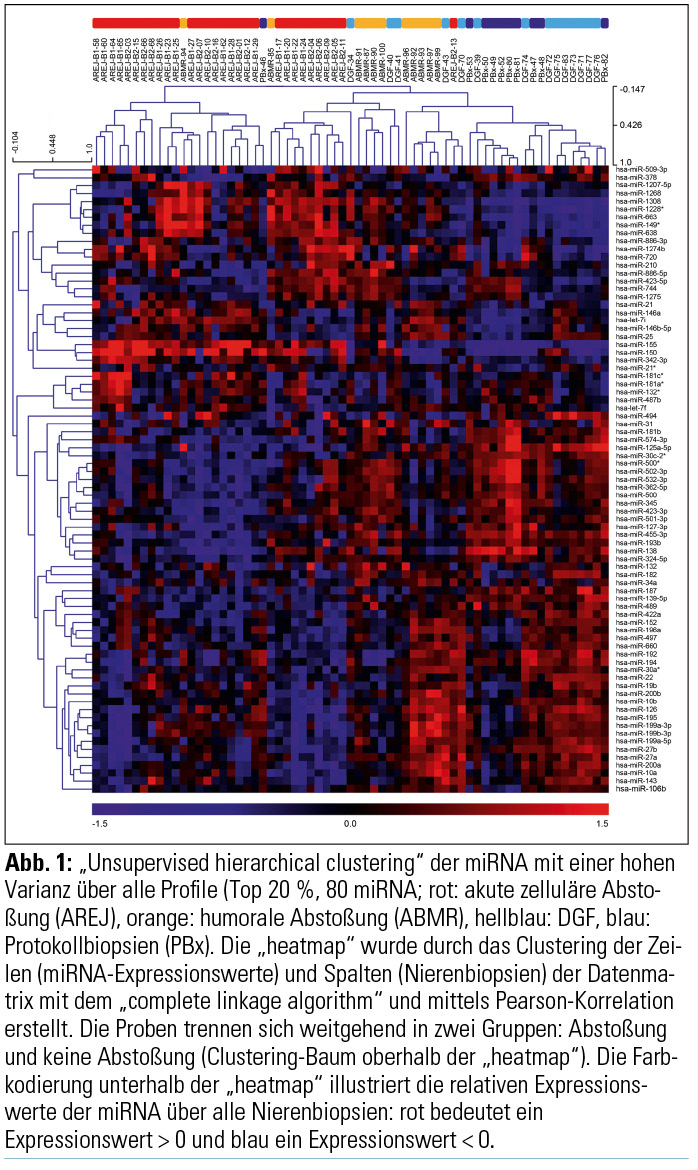
„Unsupervised hierarchical clustering“ zeigt, dass sich anhand distinkter Expressionsprofile von miRNA akute Abstoßung und DGF von der Kontrollgruppe unterscheiden lassen (Abb. 1). In der überwachten statistischen Auswertung konnten wir insgesamt 32 miRNA identifizieren, die entweder akute zelluläre Abstoßung, humorale Abstoßung oder DGF klassifizieren können. Um eine funktionelle Aussage über diese miRNA treffen zu können, ermittelten wir die experimentell verifizierten (miRTarBase) und prädiktierten Target-Gene (DIANAmT, TargetScan und miRanda). Basierend auf diesen Target-Genlisten und bereits bestehenden mRNA-Profilen1–5 wurden in einem integrativen systembiologischen Ansatz assoziierte molekulare Signalwege für akute zelluläre und humorale Abstoßung und für DGF identifiziert. Betroffene Signalwege bei DGF sind Angiogenese, Apoptose, TGF-beta, Endothelin, VEGF- und PDGF-Signalweg. Analog konnten wir für akute Abstoßung EGF-Rezeptor, „inflammation by chemokine and cytokine“, T-Zell-Aktivierung, B-Zell-Aktivierung und „cytoskeletal regulation by Rho-GTPase“ als beteiligte Signalwege identifizieren. Der „Inflammation by chemokine and cytokine“-Signalweg und die T-Zell- und B-Zell-Aktivierung sind zentrale Wege in der Pathologie der akuten Abstoßung5, und die Identifikation dieser Pathways demonstriert die Nutzbarkeit unseres kombinatorischen Ansatzes, der miRNA-Profile, miRNA-Target-Genlisten und mRNA-Expressionsprofile integriert. Weiters wurde in unserer Forschungsgruppe ein auf JAVA basiertes bioinformatisches Tool entwickelt, welches in Kürze auf unserer Homepage zugänglich sein wird. Anhand dieses Tools wird es möglich sein, eine Liste an miRNA oder einzelne miRNA vorzulegen und die gewünschte Kombination aus Algorithmen, mit dem die Zielgene vorhergesagt werden, auszuwählen. Als Ergebnis stellt das Tool eine Liste an Zielgenen und betroffenen Pathways (KEGG oder PANTHER) zur Verfügung.
Unsere Studie zeigt erstmalig, dass klinisch pathologische Entitäten in der Spenderniere durch miRNA-Profile klassifiziert werden können. Mithilfe der Systembiologie konnten unique prozessassoziierte Pathways identifiziert werden. Diese Ergebnisse können die Grundlage für weitere miRNA-Studien in Richtung diagnostischer, prognostischer und therapeutischer Tools in der Nierentransplantation bilden.