K-ras und Mikrosatelliteninstabilität: Ursache, Nachweis, Bedeutung und Konsequenz
Was ist K-ras?
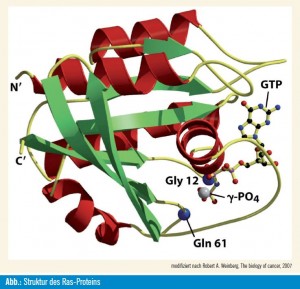 Das Onkogen K-ras ist ein Mitglied der Ras-Genfamilie. Diese Familie besteht aus 36 Genen, die für 39 Proteine kodieren. 6 Bei diesen Proteinen (z. B. p21ras) handelt es sich um kleine GTP-bindende Proteine, deren Status zwischen einer aktivierten (RAS-GTP) und einer inaktivierten Form (RAS-GDP) pendelt. Diese Proteine finden sich an der inneren Zellmembran und fungieren als Signalvermittler zwischen bestimmten Wachstumsfaktorrezeptoren der Zelloberfläche (z.B. EGFR) und ihrer nachfolgenden Signalkaskade. Auf diese Weise spielt p21ras eine wichtige Rolle in Proliferation, Differenzierung, Überleben und Transformation einer Zelle.2 Die Pathways, innerhalb derer p21ras interagiert, sind die RAF-MEK-ERK1/2-Kaskade und die PI3K-PDK1-PKB-Kaskade.5 Durch eine Mutation des regulären Gens (Wildtyp) wird K-ras zu einem Onkogen aktiviert, wodurch sich in den meisten Fällen das codierte Protein verändert (Ausnahmen bilden so genannte stumme Mutationen, bei denen das Protein in seiner Sequenz unverändert bleibt). Von dem aktiv veränderten Protein geht ein permanentes Signal aus, welches durch das enzymatische Regelwerk nicht mehr abzustellen ist. Die betroffene Zelle wird dadurch in einen stetigen Zustand einer unkontrollierten Wachstumsbereitschaft versetzt.
Das Onkogen K-ras ist ein Mitglied der Ras-Genfamilie. Diese Familie besteht aus 36 Genen, die für 39 Proteine kodieren. 6 Bei diesen Proteinen (z. B. p21ras) handelt es sich um kleine GTP-bindende Proteine, deren Status zwischen einer aktivierten (RAS-GTP) und einer inaktivierten Form (RAS-GDP) pendelt. Diese Proteine finden sich an der inneren Zellmembran und fungieren als Signalvermittler zwischen bestimmten Wachstumsfaktorrezeptoren der Zelloberfläche (z.B. EGFR) und ihrer nachfolgenden Signalkaskade. Auf diese Weise spielt p21ras eine wichtige Rolle in Proliferation, Differenzierung, Überleben und Transformation einer Zelle.2 Die Pathways, innerhalb derer p21ras interagiert, sind die RAF-MEK-ERK1/2-Kaskade und die PI3K-PDK1-PKB-Kaskade.5 Durch eine Mutation des regulären Gens (Wildtyp) wird K-ras zu einem Onkogen aktiviert, wodurch sich in den meisten Fällen das codierte Protein verändert (Ausnahmen bilden so genannte stumme Mutationen, bei denen das Protein in seiner Sequenz unverändert bleibt). Von dem aktiv veränderten Protein geht ein permanentes Signal aus, welches durch das enzymatische Regelwerk nicht mehr abzustellen ist. Die betroffene Zelle wird dadurch in einen stetigen Zustand einer unkontrollierten Wachstumsbereitschaft versetzt.
Ein weiteres Gen, dem ähnliche Bedeutung zugemessen wird, ist das B-raf- Gen. B-raf kodiert für ein Protein, das in der Kaskade dem p21ras nachgeschaltet ist und daher einen K-ras ähnlichen Effekt hat. Interessanterweise treten K-rasund B-raf-Mutationen praktisch nie im selben Tumor auf. Das heißt, dass bei einer vorliegenden K-ras-Mutation keine B-raf-Mutation zu erwarten ist und umgekehrt. Bei den sporadischen MSI-Hkolorektalen Karzinomen findet man in 34–70 % der Fälle eine Mutation des B-raf-Gens mit einer Veränderung des Codons 600 im Exon 15.3
Welche relevanten Mutationen sind bei K-ras bekannt?
Somatische Mutationen des K-ras-Gens finden sich bei vielen Tumorarten, die üblicherweise schlecht auf gängige Therapien ansprechen, wie zum Beispiel Karzinome der Lunge, des Kolons und des Pankreas.
Nachweis der K-ras-Mutation
Diagnostisch kann der K-ras-Mutationsstatus mit Hilfe der direkten Sequenzierung, einer Realtime-PCR oder mit einer Blot-Hybridisierung mit mutationsspezifischen Proben festgestellt werden. Die Hot Spots für die Mutationen betreffen die Codons 12, 13 am Exon 2 und 61 am Exon 3. Die Kenntnis der genauen Codons erleichtert den Nachweis, da nur ein kleiner Abschnitt des Gens beachtet werden muss.
Was bedeutet Mikrosatelliteninstabilität?
Die Mikrosatelliteninstabilität (MSI) weist auf eine Störung der DNA-Sequenz des Mismatch-Repair-Systems (MMR) hin. Das MMR-System kann dadurch seine Aufgabe, nämlich Fehler zu korrigieren, die bei der DNA-Replikation auftreten, nicht mehr erfüllen. Es kommt typischerweise zu einer Akkumulation von einzelnen Mutationen und Längenveränderungen in repetitiven Mikrosatelliten-Sequenzen im gesamten Genom.4 Durch die Inaktivierung einzelner MMR-Gene kommt es in der Folge zu multiplen Mutationen von Suppressor-Genen mit der daraus folgenden Inaktivierung dieser Gene und einer dadurch erhöhten Neigung zur Tumorbildung.
Wann kommt es zur Mikrosatelliteninstabilität?
Anfangs wurde die Mikrosatelliteninstabilität mit den hereditären nichtpolypösen Kolorektalkarzinomen (HNPCC) – auch unter dem Namen Lynch-Syndrom bekannt – in Verbindung gebracht. So konnte in 90 % der Fälle von HNPCC eine MSI nachgewiesen werden. Jedoch findet sich in 20–30 % der Fälle von sporadischen Kolorektalkarzinomen eben – falls eine Mikrosatelliteninstabilität (MSI).4 Die Ursache in den meisten sporadischen Fällen einer MSI liegt im so genannten Silencing der Promoterregion der Mismatch-Reparaturgene (MLH1, MSH2, MSH6) durch Hypermethylierung, wodurch das Gen inaktiviert wird.7 Beim HNPCC kommt es zu einer Keimbahnmutation in einem von fünf Mismatch- Reparaturgenen (vorwiegend in MLH1, MSH2 sowie seltener in MSH6, PMS2 und PMS1).8 Tumoren mit einem hohen Level an MSI (MSI-H) kommen typischerweise in jüngeren Jahren vor (Altergipfel < 60 Jahre), assoziiert mit einem fortgeschrittenerem T-Stadium. Die bevorzugte Lokalisation ist der rechte Darmabschnitt. Die MSI-H-Tumoren zeigen auch ungewöhnliche histopathologische Charakteristika wie etwa muzinöse Differenzierung oder niedrigen Differenzierungsgrad (G3). Ein wichtiger Unterschied zwischen dem MSI-H- und MSIstable- Status der Karzinome ist deren prognostische Bedeutung.1 Popat et al. zeigte in einem Review von 32 Studien, dass der MSI-H-Status mit einer deutlich besseren Prognose assoziiert ist als der MSI-stable-Status.9
Nachweis der Mikrosatelliteninstabilität
Zum Nachweis einer MSI sollte zuerst eine Immunhistochemie durchgeführt werden, um eine Präsenz von MLH1-, MSH2-, MSH6- und PMS2-Proteinen nachzuweisen. Als MSI-H-positiv gilt ein Tumor, wenn weniger als 10 % der Tumorzellen eine positive Reaktivität aufweisen. Eine weitere Methode ist der Nachweis der mutierten DNA mit Hilfe einer Primer-spezifischen Realtime- PCR.10 Als MSI-H werden Tumoren definiert, die an _ 50 % der gescreenten Loci eine Instabilität aufweisen; als MSILow, wenn wenigstens 1, aber _ 50 % der Loci betroffen sind; und MSS (microsatellite stable) wenn keine einzige Instabilität nachweisbar ist.11 Als Mikrosatellitenmarker werden entsprechend einer NCI-Empfehlung fünf Marker verwendet (Mononukleotidmarker BAT25 und BAT26; Dinukleotidmarker D2S123, D5S346 und D17S250).10 Unabhängig davon scheint auch ein Test von drei Markern ausreichend zu sein (BAT25, BAT26 und D2S123), wie von Iyare Esemuede et al. vorgeschlagen wurde.12
1 Winder et al., Molecular predictive and prognostic markers in colon cancer. Cancer Treat Rev 2010, doi:10.1016/j.ctrv.2010.03.005
2 Downward J et al., Prelude to an Anniversary for the RAS Oncogene. Science Vol 314.20. October 2006
3 Minoo P et al., Role of BRAF-V600E in the serrated pathway of colorectal tumourgenesis.J Pathol 2007; 212:124–33
4 Peltomaki P, Role of DNA mismatch repair defects in the pathogenesis of human cancer. J Clin Oncol 2003; 21:1174–9
5 Markowitz SD et al., Molecular Basis of Colorectal Cancer. N Engl J Med 2009; 361:2449–60
6 Karnoub AE, Ras oncogenes: split personalities. Nature Reviews Molecular Cell Biology vol 9 (July 2008):517–531
7 Cunningham JM et al., Hypermethylation of the hMLH promotor in colon cancer with microsatellite instability. Cancer Res 1998; 58:3455–60
8 Nash GM et al., Automated, multiplex assay for high frequency micro – satellite instability in colorectal cancer. J Clin Oncol 2003; 21:3105–12
9 Popat S et al., Systematic review of Microsatellite Instability and colorectal Cancer prognosis. J Clin Oncol 2005; 23:609–18
10 Boland CR et al., A National Cancer Institute Workshop on Microsatellite Instability for cancer detection and familial predisposition: development of international criteria for the determination of microsatellite instability in colorectal cancer
11 Bertagnolli MM et al., Microsatellite instability predicts improved response to adjuvant therapy with irinotecan, fluorouracil and leucovorin in stage III colon cancer: Cancer and Leukemia Group B Protocol 89803. J. Clin Oncol 2009; 27:1814–21
12 Iyare Esemuede et al., Improved Testing for Microsatellite Instability in Colorectal Cancer using a simplified 3-Marker Assay. Ann Surg Oncol 2010; 17:3370–3378