Gensignaturen beim Mammakarzinom
Hintergrund, Entwicklung
Von den jährlich 5.000 Frauen, die in Österreich jedes Jahr an einem Mammakarzinom erkranken, würde ohne adjuvante medikamentöse Therapie ungefähr jede dritte Frau nach der Ersterkrankung ein Rezidiv erleiden und an ihrer Erkrankung versterben. Welche Patientin nun ein höheres oder niedrigeres Rezidivrisiko hat, wurde bislang routinemäßig mittels klinisch-pathologischer Eigenschaften wie Alter, Tumorgröße, Grading oder Nodal- und Hormonrezeptorstatus bestimmt. Diese Faktoren wurden dann zusammengefasst und in Risikokategorien eingeteilt, wie zum Beispiel in den St.-Gallen-Konsensus-Empfehlungen bis 2009 oder in der AdjuvantOnline®- Software. Bei diesem frei erhältlichen Computerprogramm werden die klinischpathologischen Parameter und die geplante adjuvante Therapie eingegeben, und der Algorithmus berechnet das Rezidiv- bzw. Mortalitätsrisiko einer einzelnen Patientin. Die Berechnung basiert auf den Ergebnissen der amerikanischen SEER-Datenbank (Surveillance, Epidemiology and End Results), wo seit 1973 Daten des National Cancer Institute (NCI) gesammelt werden. Eine weitere Möglichkeit ist die Verwendung von Nomogrammen wie dem so genannten Milan- Nomogramm (Colleoni et al., Eur J Cancer 2010), wobei Hormonrezeptoren und der Proliferationsmarker Ki-67 mit der geplanten Zahl der Zyklen einer neoadjuvanten Therapie kombiniert werden, um das Ansprechen auf diese vorhersagen zu können.
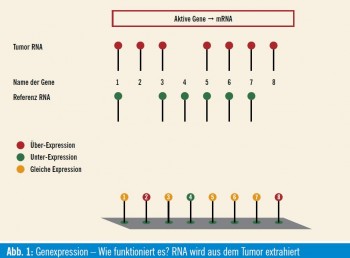
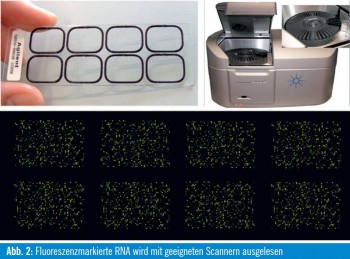
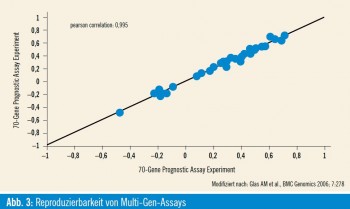
Diese Einschätzungen waren jedoch oft ungenau, und wir konnten das individuelle Risiko der einzelnen Patientin nur sehr unzureichend vorhersagen. Einige prognostische und prädiktive Faktoren wie Hormonrezeptorstatus und HER2- Expression des Tumorgewebes werden routinemäßig bestimmt und haben bereits seit Langem Einfluss auf die Behandlung. Durch den Einsatz neuer Techniken wie z. B. quantitative Realtime- PCR oder RNA-Microarrays ist es in den letzten Jahren möglich geworden, molekulare Profile individueller Patientinnen zu erstellen und so die Über- oder Unterexpression tausender tumorspezifischer Gene gleichzeitig in einem Experiment zu untersuchen. Dabei wird RNA aus dem Tumor extrahiert und gemeinsam mit Referenz-RNA auf einen Objektträger aufgebracht (Abb. 1). Die Farbintensität der fluoreszenzmarkierten RNA wird mit geeigneten Scannern ausgelesen und von einem Computerprogramm interpretiert (Abb. 2). Bestimmte Gruppen von Genen können in so genannten Profilen von diesen Microarrays ausgelesen werden und erlauben so die Bestimmung z. B. der Prognose einer Mammakarzinompatientin oder des Ansprechens auf eine adjuvante Therapie. Beim Test eines einzelnen Biomarkers, wie zum Beispiel EGFR (Epidermal Growth Factor Receptor), der zwar breit und kostengünstig verfügbar ist, tritt aber eine hohe Rate an so genannten screening failures auf. Das heißt, dass viele Patientinnen von einer zielgerichteten Therapie auf diesen einen Biomarker nicht profitieren, weil viele relevante molekulare Veränderungen im Tumor nicht erfasst werden. Multi-Gen- Assays hingegen sind zwar noch nicht überall verfügbar, aber screening failures werden minimiert, da sie keinen Test für ein einzelnes Target darstellen. Außerdem sind diese Genprofile deutlich besser in verschiedenen Labors und zu verschiedenen Zeitpunkten reproduzierbar als herkömmliche immunhistochemische Färbungen (Abb. 3).
Klinisch verfügbare Profile
70 Gene, MammaPrint®: Für den klinischen Alltag sind seit mehreren Jahren vor allem zwei Gensignaturen verfügbar. Das 70-Gen-Profil MammaPrint® ist seit Kurzem nicht nur aus frischem oder gefrorenem Tumorgewebe verfügbar, sondern kann nun auch aus Paraffinmaterial die Prognose einer Patientin bestimmen. Dabei wird das Rezidivrisiko einer individuellen Patientin entweder als Low Risk oder High Risk in 2 Kategorien eingeteilt. Bei der Auwertung dieses 70-Gen-Profils werden aber nicht nur die 70 Gene für das Rezidivrisiko mehrfach und höchst reproduzierbar ausgewertet, sondern auch die Hormonrezeptoren und der HER2-Status werden auf Ebene der Genexpression routinemäßig mitbestimmt und auf Wunsch auch die Expression von 56 Forschungsgenen wie z. B. BRCA1 und -2, VEGF, KRAS, mTOR, EGFR, IGFR, PIK3CA und viele mehr.
21 Gene, oncotype DX®: Der 21-Gen- Recurrence-Score oncotype DX® verwendet die Technik der Realtime-PCR, wobei durch Bestimmung von 16 tumorspezifischen und 5 Kontrollgenen die Patientinnen in 3 Risikokategorien – Low Risk, Intermediate Risk und High Risk – eingeteilt werden. Dieses Profil wurde in mehreren zuvor durchgeführten prospektiven Studien retrospektiv validiert und gibt ein Rezidivrisiko für die Patientin in einem Score von 0–100 an. Beide Profile wurden auch für lymphknotenmetastasierte Patientinnen untersucht und haben gezeigt, dass bei Vorliegen von wenigen Lymphknotenmetastasen die Biologie des Primärtumors viel entscheidender für die Prognose der Patientin ist als das Vorliegen von positiven Lymphknoten. Die Lymphknotenmetastasierung stellt eher ein Zeichen der Tumorlast dar als eine aggressivere Biologie, was auch die Theorie unterstützt, dass es sich bei der chirurgischen Axilladissektion meistens um ein reines Staging-Verfahren handelt und nicht um ein tumorspezifische Therapie, die die Prognose unserer Patientinnen verbessern kann.
EndoPredict®: Viele weitere Genexpressionsprofile wurden in den letzten Jahren entwickelt, wie zum Beispiel der Genomic Grade Index (GGI) zur verlässlichen Bestimmung der Proliferation eines Tumors oder das Profil EndoPredict®, welches zusammen mit der österreichischen Studiengruppe ABCSG entwickelt und an Patientinnen aus diesen Studien validiert wurde. Für die beiden erstgenannten Profile
(MammaPrint®, oncotype DX®) wurden in den letzten Jahren zwei große prospektiv randomisierte Studien in Europa und den USA durchgeführt (MINDACT und TAILORx). Die Einbringung mehrerer tausend Patientinnen in diese adjuvanten Studien konnte im Jahr 2011 abgeschlossen werden, allerdings werden die ersten Ergebnisse noch einige Jahre auf sich warten lassen.
Neue Ergebnisse und Zukunftsperspektive
Bei der European Breast Cancer Conference (EBCC 8) in Wien, wo ca. 5.000 Teilnehmer ein interessantes Programm verfolgen konnten, wurden auch im Bereich der Gensignaturen interessante Studien präsentiert.
RASTER-Studie (NO-Patienten): Als Highlight wurden zum ersten Mal prospektive 5-Jahres-Daten für Mamma- Print® präsentiert. In der so genannten RASTER-Studie wurden zwischen 2004 und 2006 in 16 verschiedenen öffentlichen Krankenhäusern jeder Größe in den Niederlanden 427 lymphknotennegative Patientinnen mit dem 70-Gen-Profil untersucht. Mit Hilfe des Genprofils wurden 51 % aller Patienten als Low Risk für Fernmetastasen klassifiziert. Die anschließende Therapie wurde je nach klinischer und genomischer Risikoeinschätzung den behandelnden Ärzten und den Patientinnen überlassen. Der Test des 70-Gen-Profils erfolgte gegen das Computerprogramm Adjuvant! Online (AOL) und interessanterweise wurde aufgrund der damals gültigen, sehr restriktiven Therapie-Guidelines in den Niederlanden ein großer Teil der Patientinnen (39 %) ganz ohne adjuvante medikamentöse Therapie behandelt. Dies ist für die korrekte Evaluation eines prognostischen Tests von ausgesprochener Wichtigkeit, um hier eine Vermischung mit der Prädiktion auf die durchgeführte Therapie zu vermeiden. Die Studie bestätigte den zusätzlichen prognostischen Wert des Genprofils über die Einschätzung mittels klinisch-pathologischer Risikoparameter hinaus. Die Patienten mit einem Low-Risk-Genprofil, aber einem hohen Rezidivrisiko mittels AOL – also mit diskordanter Risikoeinschätzung – hatten ein metastasenfreies 5-JahreÜberleben von 100 %. Somit könnte bei zusätzlich 29 % der Patientinnen mit ausreichender onkologischer Sicherheit auf eine adjuvante Chemotherapie verzichtet werden. Dies ist auch im klinischen Alltag die Gruppe von Patientinnen, die von einer zusätzlichen Diagnostik mit einem Genprofil profitiert.
Dänische Studie (N1–3-Patienten): Die zweite interessante Studie wurde von Tramm et al. von der dänischen Studiengruppe (DBCG) präsentiert. Aus den randomisierten Studien DBCG 82bc, in denen in der viel diskutierten Gruppe von Patientinnen mit 1–3-positiven Lymphknoten nach Mastektomie eine Post- Mastektomie-Radiotherapie (PMRT) untersucht wurde, konnte von 195 eingefrorenen Tumorproben ein whole genome array durchgeführt werden. Die Patientinnen waren zwischen 1983 und 1989 behandelt worden, und es fand sich eine neue 7-Gen-Signatur, die diejenigen Patientinnen mit signifikant höherem Lokalrezidivrisiko identifizieren konnte, die dann auch von der PMRT profitierten. Das Profil zeigt also prognostischen und prädiktiven Nutzen und muss nun noch in unabhängigen Studien validiert werden, bevor es in den klinischen Alltag integriert werden kann.
PAM50 Breast Cancer Intrinsic Classifier ®: Auch Dr. Charles Perou von der University of North Carolina at Chapel Hill gab im Eröffnungssymposium des EBCC am Mittwoch einen Überblick über die Entwicklung der intrinsischen Subtypen des Mammakarzinoms. Aufgrund der Daten aus Genexpressionsexperimenten konnte er zeigen – was inzwischen breit anerkannt ist –, dass das Mammakarzinom keine einheitliche Erkrankung ist. Vielmehr haben wir es mit einem Spektrum an Subtypen zu tun mit klarer Zuordnung zu verschiedener zellulärer Abstimmung. Die Begriffe Luminal A/B, HER2-Subtyp, basal-like und Claudin- low haben mittlerweile auch therapeutische Relevanz in den St.-Gallen- Konsensus-Richtlinien von 2011 erhalten. Es gibt hier markante Unterschiede in Bezug auf Inzidenz, Überleben, Ort der Metastasierung und Ansprechen auf adjuvante Therapie. Die Forscher um Perou haben nun auch einen Test entwickelt, der aus paraffinfixiertem Material diese Subtypen durch Genexpression von 50 Genen bestimmen kann (PAM50 Breast Cancer Intrinsic Classifier®) und nun klinisch evaluiert wird.
Next generation sequencing: Dr. Christos Sotiriou vom Institut Jules Bordet in Brüssel lieferte einen Einblick in die Zukunft des so genannten next generation sequencing. Hier werden Techniken wie „Exome sequencing“ auf uns zukommen, wobei verschiedene Mutationsmuster im gesamten Genom eines Menschen untersucht werden. Was beim human genome project mit der Sequenzierung aller 2,9 Milliarden Basenpaare des menschlichen Genoms noch von 1990–2001 gedauert und über 1000 Wissenschaftler beschäftigt hat, ist nun mittels next generation sequencing in 2 Wochen möglich geworden. Man erwartet sich zum Beispiel, aus der Heterogenität von Tumoren Rückschlüsse auf Prognose und Prädiktion tätigen zu können. Mit Techniken wie dem RNA sequencing zur Erforschung der Aktivierung verschiedener Pathways und DNA rearrangements zur Bestimmung der minimalen Resterkrankung im Plasma von Patientinnen könnten neue therapeutische Strategien entwickelt werden. So ist eine fast unüberschaubare Menge an neuen Techniken zu erwarten, die in den nächsten Jahren teilweise in die Klinik Einzug halten werden, und wir werden lernen, diese zum besten Nutzen für unsere Patientinnen einzusetzen.